日本ジェネティクスのアプリケーションノートとは?
- 当社製品を実際にご使用頂いた、正真正銘、日本国内の研究者様による評価データ
- 製品をご検討中の方はもちろん、すでにお使いのお客様におかれましても、類似の研究をされている他の研究者の方の事例集としてご活用頂けます
- アプリケーション検索専用ページはこちら
| アプリケーションノート 2018<04> 製品名: MyTaq DNA Polymerase(BIO-21105) メーカー名: Bioline 社 |
下記データは、栃木県農業試験場 研究開発部 生物工学研究室 生井 潔様の御厚意により掲載させていただきました。
本アプリケーションノートでは、MyTaq DNA Polymeraseによりニラ単為発生性連鎖マーカーおよび複相大胞子形成性連鎖マーカーをそれぞれPCR増幅し、単為生殖性の識別に応用している事例をご紹介します。
はじめに
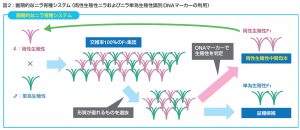
ニラは、交配しても数%の個体にしか父親の遺伝子が伝わらず、残りの個体は母親と全く同じ遺伝子型となります(図1)。
この性質を単為生殖性といい、父親の遺伝子が伝わる確率(交雑率)は交配に用いる母親によって異なります。
この単為生殖性は、種子生産を行うには都合の良い性質ですが、交配しても得られる個体のほとんどが母親と同じ遺伝子型となるため、新品種を開発するには不都合な性質です。
これまでの研究から、単為生殖性を持たない(両性生殖性)ニラの開発に成功し、さらに単為生殖性か両性生殖性かを識別する DNA マーカーを開発しました(図2)。
なお、ニラの単為発生性は、単為発生因子と複相大胞子形成因子との2つの因子により構成されるため(diplospory 型)、2つの連鎖マーカーの検出が必要になります。
単為発生因子はPCR後の電気泳動による目的バンド(マーカーA:148bp)の有無、複相大胞子形成因子はPCR後のフラグメント解析による目的ピーク(マーカーB:267nt)の有無で確認しました。

【参考文献】
1) 中澤佳子・生井 潔・小島昭夫・小林俊一・田﨑公久・天谷正行(2006)四倍体ニラにおける単為発生性の遺伝様式.育種学研究 8: 89-98.
2) 天谷正行・中澤佳子・松本紀子・飯村一成(2010)バルクセグレガント法によるニラ四倍体(Allium ramosum, syn. A.tuberosum 2n=4x=32)の単為発生
性連鎖マーカーの開発 育種学研究 12: 73-80
方法と結果
• 初発サンプルの種類と量:葉身 約3 ~ 5mg(幅1mm強×5 ~ 6mm程度)
• DNA抽出方法:TPS法(下記フロー参照)
• 溶出液量:100μl
• 概ねのDNA濃度:10ng/μl
• 単為生殖性識別DNAマーカー:
① マーカーA:単為発生性連鎖マーカー
② マーカーB:複相大胞子形成性連鎖マーカー
ニラセル苗における簡易DNA抽出法(TPS法)
① 96穴PCRプレートにステンレスビーズを入れる(1穴:2個)。
② TPSバッファー[10mM EDTA(pH8.0), 1M KCl, 100mM Tris-HCl(pH8.0)]
を125μlずつ加える
③ ピンセットで葉身(5mm×1mm程度、重さ約3 ~ 5mg)をサンプリングする。
④ 96穴PCRプレート用マット(液が漏れない物)で蓋をする。
⑤ ビーズクラッシャーで破砕する。
⑥ 4,800rpmで10 分間遠心する。
⑦ 上清50μlとり、新しい96穴PCRプレートに移す。
⑧ イソプロパノール50μlを加えて混和する。
⑨ 4,800rpmで20 分間遠心した後、上清を捨てる。
⑩ 10 分間エバポレーターで乾燥させる。
⑪ TE・RNase 100μlを加え、37℃で30 分間インキュベートする。
ニラ生殖性判定
① 単為発生性連鎖マーカー(マーカーA)
1)PCR反応液組成
5x MyTaq Reaction Buffer 2.0 μl
primerA-F(10μM) 0.333 μl
primerA-R(10μM) 0.333 μl
primerPC-F(10μM) 0.067 μl ※PC:ポジティブコントロール
primerPC-R(10μM)) 0.067 μl
MyTaq DNA Polymerase 0.05 μl
DW 6.15 μl
DNA溶液 1.0 μl
———————————————
Total 10 μl
2)PCR装置:GeneAmp®PCR system9700(Applied Biosystems)
3)PCRプログラム
95℃ 3分
95℃ 15 秒
61℃* 15 秒 ×10サイクル(*-0.2℃/サイクル)
72℃ 15 秒
95℃ 15 秒
59℃ 15 秒 ×25サイクル
72℃ 15 秒
72℃ 30 秒
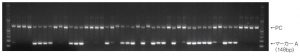
4)増幅サイズ
単為発生性連鎖マーカーA(PLM4):148bp
PC:約590bp(GelRedによるプレステインのため、泳動像ではサイズが大きく見える)
5)電気泳動条件
マーカー:100bp DNA ladder
1xTBE buffer、1.5% agarose gel、3/100,000 量のGelRedTM Nucleic Acid Gel Stain(コスモバイオ社)、100v、45min.、
PCR産物5μlをアプライ
6)電気泳動像
② 複相大胞子形成性連鎖マーカー(マーカーB)
1)PCR反応液組成
5x MyTaq Reaction Buffer 2.0 μl
primerB-M13Rv-F(0.4μM) 1.0 μl
primerB-R(1.6μM) 1.0 μl
蛍光primerM13Rv-Dye4(8μM) 0.1 μl
MyTaq DNA Polymerase 0.05 μl
DW 4.85 μl
DNA溶液 1.0 μl
———————————————————–
Total 10 μl
2)PCR装置:GeneAmp®PCR system9700(Applied Biosystems)
3)PCRプログラム
94℃ 5分
94℃ 30 秒
56℃ 45 秒 ×30サイクル
72℃ 45 秒
94℃ 30 秒
53℃ 45 秒 ×8サイクル
72℃ 45 秒
72℃ 7分
4)増幅サイズ
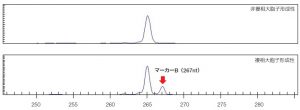
複相大胞子形成性連鎖マーカーB(nr2902_267):267nt
5)マーカー検出例
シーケンサー:GenomeLab GeXP(BECKMAN COULTER)
お客様のコメント
我々の研究室では、MyTaq DNA Polymeraseを通常使いのPCR酵素として使用しています。
本酵素を選定するに当たり、複数の酵素を比較し、増幅のしやすさや結果の安定性、操作性の良さ、価格等について考慮して総合的に判断しました。パート職員もPCRを実施するため、比較的熟練度が低い人でも安定して結果が得られることが重要となりますが、これまで問題無く、満足した結果が得られています。
- こちらのアプリケーションノートのPDFダウンロード : こちら
- 製品情報詳細ページ:MyTaq DNA Polymerase
- アプリケーションノート検索ページ(型番・キーワード・アプリケーションから検索可能)