日本ジェネティクスのアプリケーションノートとは?
- 当社製品を実際にご使用頂いた、正真正銘、日本国内の研究者様による評価データ
- 製品をご検討中の方はもちろん、すでにお使いのお客様におかれましても、類似の研究をされている他の研究者の方の事例集としてご活用頂けます
アプリケーション検索専用ページはこちら
| アプリケーションノート 2017<23> 製品名:KAPA HyperPlus Kit(for illumina)(KK8510, KK8512, KK8514) メーカー名:KAPA BIOSYSTEMS 社 |
下記データは、早稲田大学 ナノ・ライフ創新研究機構 細川 正人 先生のご厚意により掲載させていただきました。
概要
多量サンプルを扱うRNA-seq実験において、ライブラリ作製にかかるコストは大きく、実験規模を制約することにつながる。
多くのキットでは、推奨量として1 ng以上のインプットcDNAが必要とされている。ところが、シングルセルRNA由来のcDNAなどでは、cDNA収量がわずかであり、貴重なサンプル消費が懸念材料となる。
コスト面とインプット量制限の課題を解消するため、1細胞用のRNA-seq(増幅cDNA)ライブラリ作製プロトコルであるSMART-seq2とKAPA HyperPlus Kitを組み合わせた改変ライブラリ作製方法を検討し、他社製品と比較した。
実験条件と実験手順
細胞から抽出した50 pgのtotal RNAを用い、SMART-seq2(Nature Protocols 9,171-181(2014))の手法によりcDNAを調製した。それを用い、インプットcDNA量やLigationおよびAmplificationの反応液量を改変した、いくつかの条件でライブラリー調製を行った。
• 生物種: ヒトがん細胞株
• 初発サンプル量: total RNA 50 pg
• RNA抽出方法: RNeasy mini kit(Qiagen社) DNase I 処理実施
• cDNA調製: SMART-seq2(Nature Protocols 9,171-181(2014))
• ライブラリ調製:
KAPA HyperPlus Kit(ライゲーションベース)I 社キットN(タグメンテーションベース)
• シーケンサー: Miseq
KAPA HyperPlus 反応条件
| サンプル ID |
プロトコール | インプットcDNA | アダプター (nM) |
グループ | ||
|---|---|---|---|---|---|---|
| 濃度 (ng/uL) |
液量 (uL) |
cDNA量 (ng) |
||||
| S01 | スタンダード (推奨法) |
0.2 | 5 | 1 | 300 | K1 |
| S02 | ||||||
| S03 | ||||||
| S04 | L15_A12 | 0.2 | 1 | 0.2 | 1500 | K2 |
| S05 | ||||||
| S06 | ||||||
| S07 | 1 | 1 | 1 | K3 | ||
| S08 | ||||||
| S09 | ||||||
| S10 | 0.2 | 1 | 0.2 | 15000 | K4 | |
| S11 | ||||||
| S12 | ||||||
| S13 | 1 | 1 | 1 | K5 | ||
| S14 | ||||||
| S15 | ||||||
| S16 | L15_A15 | 0.2 | 1 | 0.2 | 1500 | K6 |
| S17 | ||||||
| S18 | ||||||
| S19 | 1 | 1 | 1 | K7 | ||
| S20 | ||||||
| S21 | ||||||
KAPA HyperPlus kit
Input DNA量:0.2 ng 1.0 ng
反応液量: L15_A12:Ligation 1/5量 Amplification 1/2 量で反応
L15_A15:Ligation 1/5量 Amplification 1/5 量で反応
増幅サイクル数:全条件15 サイクル
I 社キットN 反応条件
| サンプル ID |
インプット cDNA(ng) |
反応液量 | グループ |
|---|---|---|---|
| N.S01 | 1 (推奨法) |
1x | N1 |
| N.S02 | |||
| N.S03 | |||
| N.S04 | 0.25 | 0.25x | N2 |
| N.S05 | |||
| N.S06 | |||
| N.S07 | 0.5 | N3 | |
| N.S08 | |||
| N.S09 | |||
| N.S10 | 1 | N4 | |
| N.S11 | |||
| N.S12 |
I 社キットN
Input DNA量:0.25 ng 0.5 ng 1.0 ng
反応容量:1/4 量で反応
増幅サイクル数:全条件12 サイクル
データベースと解析ツール
| 項目 | バージョン |
|---|---|
| リファレンスゲノム | Ensembl Human Genome,Release 90 |
| アノテーションファイル | Ensembl Human GTF, Release 90, GTF |
| QC, フィルタリング | flexbar 2.4, fastq-mcf, FastQC 0.11.2 |
| ゲノムマッピング | HISAT2 2.0.5 |
| 遺伝子発現定量 | RSEM 1.3.0 |
| マッピング領域同定 | bedtools 2.26.0 |
| カバー率計算 | RSeQC(bam2wig.py) |
結 果
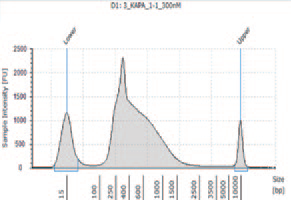
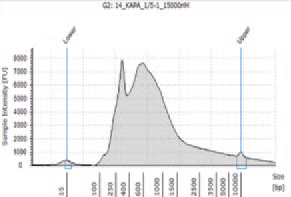
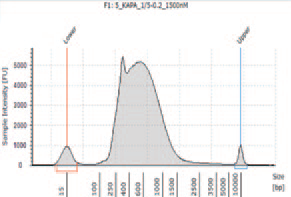
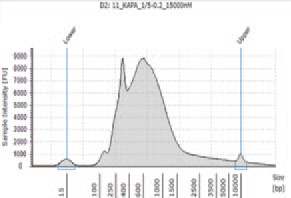
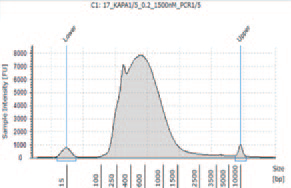
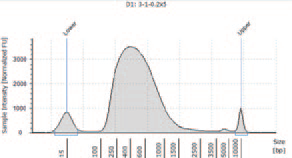
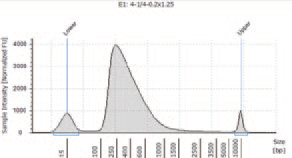
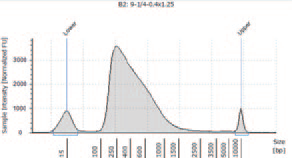
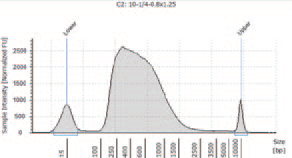
1.TapeStation によるライブラリーのサイズ確認
① KAPA HyperPlus を用いて作製したライブラリー
② I 社キットN を用いて作製したライブラリー
KAPA HyperPlus で250 bp 付近にピークが見られるが、シーケンスにおいて特に問題とはならなかった。
2.シーケンス解析データ
※下部の「各解析データ」を参照ください。
まとめ
| 項目 | KAPA HyperPlus 結果 |
I 社キットN 結果 |
評価 |
|---|---|---|---|
| シーケンス精度 | Phred Score 38 にピーク、ただしその割合は I 社キットN より少ない | Phred Score 38 にピーク | KAPA ≦ I 社キットN |
| ミスマッチ | 約12% | 約8% | KAPA < I 社キットN |
| 挿入 | 約0.3% | 約0.3% | KAPA = I 社キットN |
| 欠損 | 約0.5% | 約0.5% | KAPA = I 社キットN |
| GC 含量 | 60%にピーク | 50~ 60%にピーク (コントロールと条件変更サンプルでプロファイルが異なる) |
KAPA > I 社キットN |
| シーケンスリード長 | cDNA 増幅アダプタ配列を含むためピークが複数※1 | cDNA 増幅アダプタがほとんど検出できないためピークは1つ※1 | KAPA > I 社キットN |
| ゲノムマップ率 | 40~ 60% (マップツールのパラメータで改善可能) |
70~80% | KAPA ≦ I 社キットN |
| インサートサイズ | 150~200 bp | 50~100 bp | KAPA > I 社キットN |
| 転写物カバー率 | 全体的に一定 | 3’側に偏る | KAPA > I 社キットN |
| 検出遺伝子数 | 約10,000 | 約10,000 | KAPA = I 社キットN |
| データ再現性 | 条件によって再現性が低い時がある | 条件が同じならば再現性は高い | KAPA < I 社キットN |
| 条件変化に対する 堅牢性 |
条件が変化してもあまり変化しない | 発現量が高いとばらつく | KAPA > I 社キットN |
| ばらつき | CV 上限:0.25 (1000 TPM) CV 上限:0.5 (100 TPM) CV 上限:1.0 (10 TPM) |
CV 上限:0.25 (1000 TPM) CV 上限:0.5 (100 TPM) CV 上限:1.5 (10 TPM) |
KAPA = I 社キットN (低発現遺伝子以外) KAPA < I 社キットN (低発現遺伝子) |
| 試験濃度依存性 | 濃度を変化させてもほぼコントロールと同等 | 濃度変化させると、高発現遺伝子の再現性がやや低下 GC 含量によって発現量の変化大 |
KAPA > I 社キットN (再現性) KAPA > I 社キットN (GC 含量による発現量の変化) |
※1(補足)
シーケンスリード長は、SMART-seq2 で使用しているcDNA 化およびPCR 時のアダプター配列を除去したあとのリード長を解析した。
したがって、特に短いリードは、もともとはPCR 後のアンプリコン末端を含んでいた配列であった。
KAPA HyperPlus はライゲーションベースのため、これらの配列がピークとして出て来ていると推察される。
一方、I 社キットN は、タグメンテーションベースのため、アンプリコン末端は原理的にリードとしてデータに出て来ないと推察される。
■ 総評
特に以下の点が評価され、KAPA HyperPlus のほうが優れていると判断された。
条件としてK6(Ligation1/5 量、Amplification1/5 量、インプットcDNA0.2 ng)でも運用可能と判断された。
• GC 含量による偏りが小さい
• インサートサイズが長い。

各解析データ
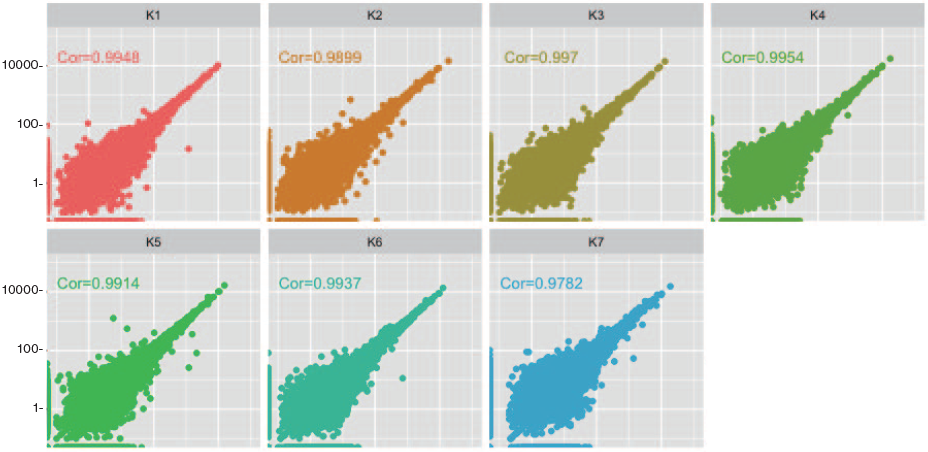
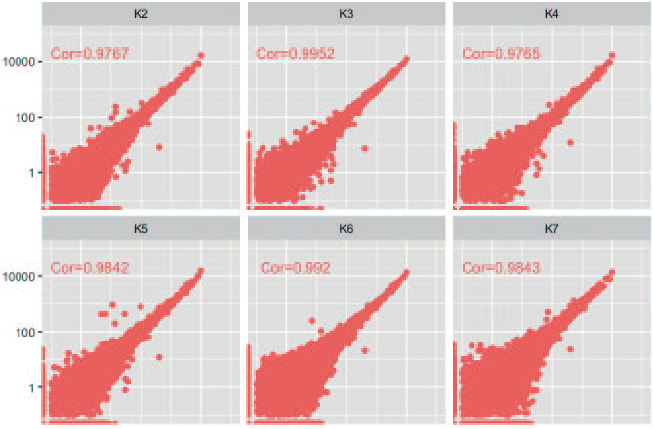
1.遺伝子発現定量の再現性
• 同条件でのサンプル間のばらつきを比較
• K7 を除き、いずれの相関係数も 0.99 以上で、ばらつきはほぼなかった。
KAPA HyperPlus
I 社キットN
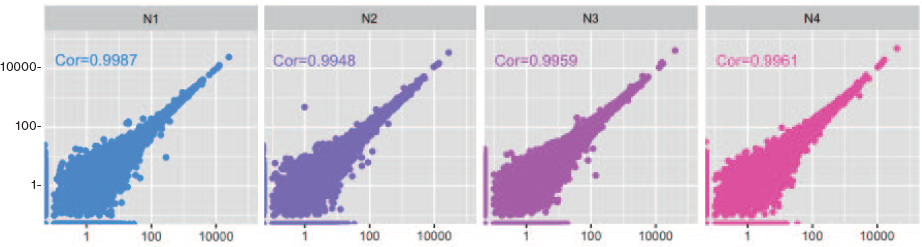
2. コントロールとの比較
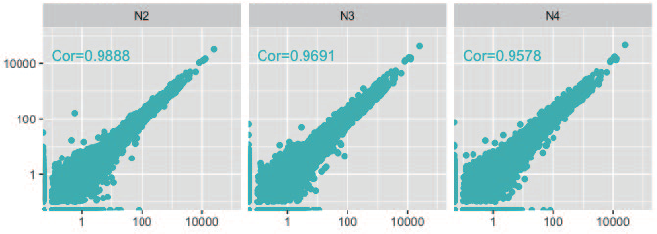
• K1, N1 をコントロールとして、再現性を比較
• KAPA HyperPlus ではコントロールとの整合性が高かった。
• I 社キットN は、N2 を除き、高発現領域の遺伝子の再現性が低かった。
KAPA HyperPlus
I 社キットN
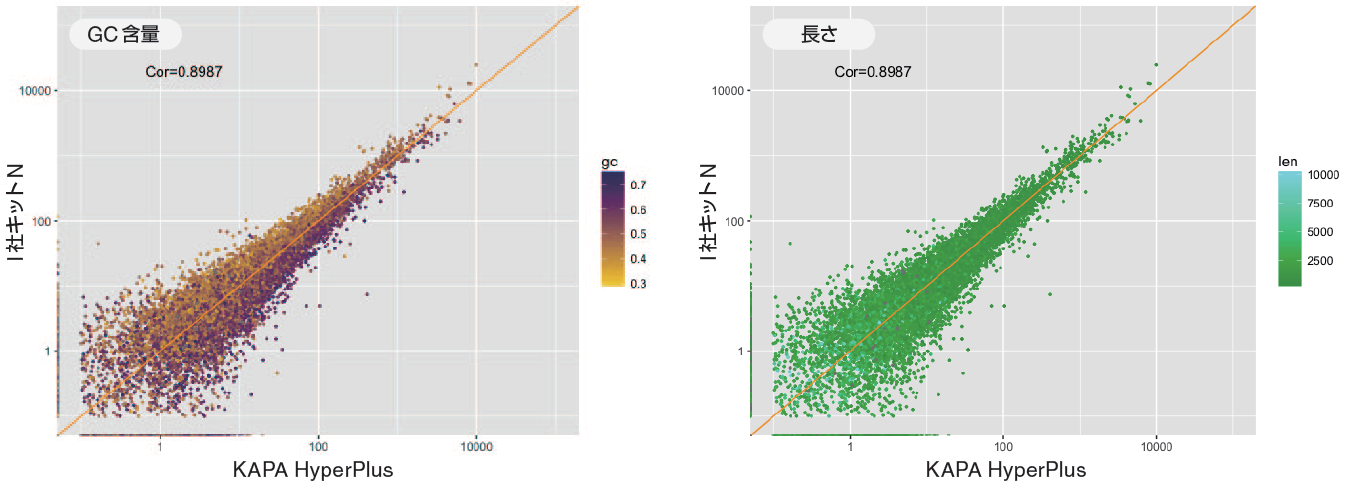
3.KAPA HyperPlus とI 社キットN の比較(GC 含量および長さの影響)
• K1(KAPA HyperPlus)とN1( I 社キットN)を比較
• GC 含量によって発現量に偏りの発生が見られた。
• 一方、長さによる影響は見られなかった。
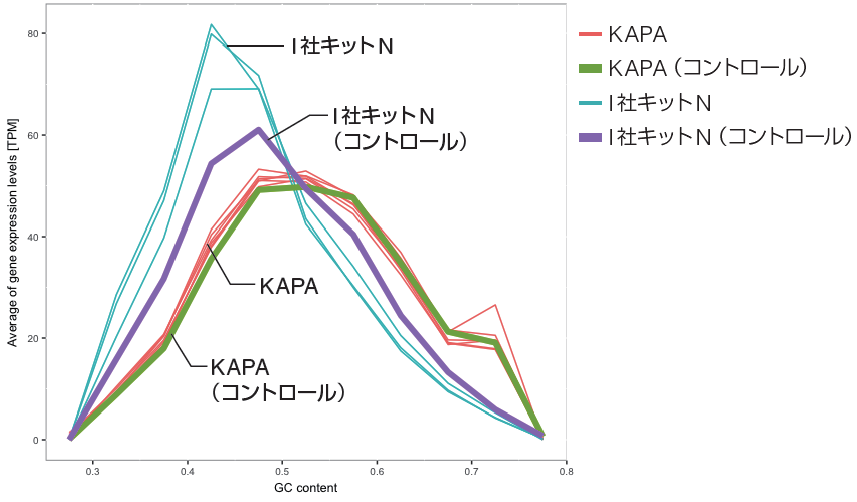
4.各サンプルのGC 含量ごとの平均遺伝子発現量
• I 社キットN はGC 含量が低い遺伝子の方に発現量に偏りが見られた。
• KAPA HyperPlus では、各条件で安定してコントロールと同じ傾向を示した。
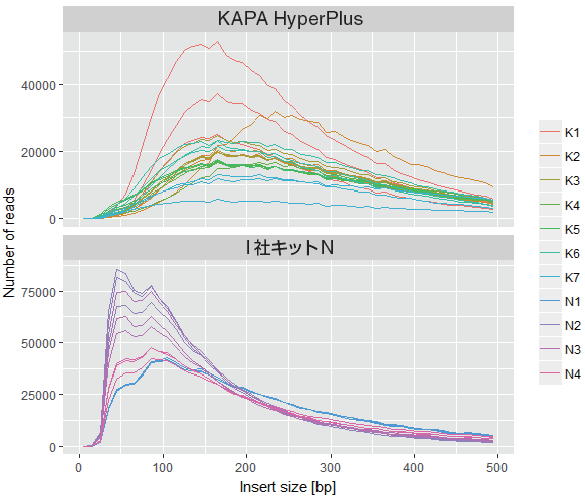
5.各サンプルのインサートサイズの分布
• マッピングの結果から、インサートサイズを計算した。
• インサートサイズは KAPA HyperPlus の方が長かった。
I 社キットN :インサートサイズ最頻値100 bp 以下
KAPA HyperPlus :インサートサイズ最頻値150 ~ 200 bp
- こちらのアプリケーションノートのPDFダウンロード : こちら
- 製品情報詳細ページ: KAPA HyperPlus Kit(for illumina)
- アプリケーションノート検索ページ(型番・キーワード・アプリケーションから検索可能)