日本ジェネティクスのアプリケーションノートとは?
- 当社製品を実際にご使用頂いた、正真正銘、日本国内の研究者様による評価データ
- 製品をご検討中の方はもちろん、すでにお使いのお客様におかれましても、類似の研究をされ粗抽出ている他の研究者の方の事例集としてご活用頂けます
アプリケーション検索専用ページはこちら
| アプリケーションノート 2017<21> 製品名:自動DNA断片ゲル抽出システム BluePippin(BLU0001) メーカー名:Sage Science社 |
下記データは、慶應義塾大学 先端生命科学研究所 荒川 和晴 様のご厚意により掲載させていただきました。
背景
一般的に、次世代シーケンスにおいては、現在のところ、短いライブラリのデータが優先的に得られやすい傾向がある。この場合、「サイズセレクション」により目的サイズを分離回収したサンプルを使用してライブラリを作製することが、より効果的に目的のデータを得るための解決策となる。
本アプリケーションノートでは、BluePippinを用いてサイズセレクションを行ったサンプルを用いて作製したライブラリを、MinION(Oxford Nanopore Technologies)でシーケンスを行った結果の一例を紹介する。
実験条件と実験手順
| ● 生物種 アメリカジョロウグモ(Nephila clavipes)の成体の脚一本 ① サンプルDNA抽出 |
 |
| ② Agilent TapeStation Genomic ScreenTapeでサイズ確認 InvitrogenTM QubitTM システムでDNA量の確認 ③ BluePippinで10 kb以上のDNAサンプルをサイズセレクション ④ Agilent TapeStation Genomic ScreenTapeでサイズ確認 ⑤ ライブラリ作製 ⑥ Agilent TapeStation Genomic ScreenTapeでサイズ確認 ⑦ MinION(Oxford Nanopore Technologies)によるシーケンス解析 |
|
 |
 |
結果
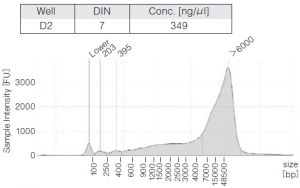
| ② Agilent TapeStation Genomic ScreenTape によるサイズ確認 (BluePippin前) |
 |
 |
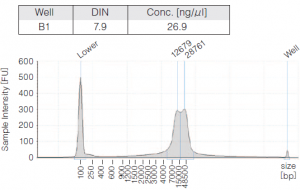
| ② Agilent TapeStation Genomic ScreenTape によるサイズ確認 (BluePippin後) |
 |
 |
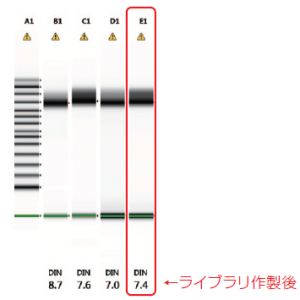
| ⑥ Agilent TapeStation Genomic ScreenTape によるサイズ確認 (ライブラリ作製後) |
 |
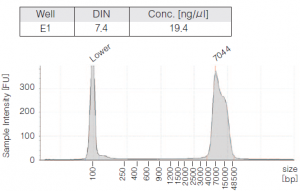 |
| ⑥ シーケンスの結果 | |||
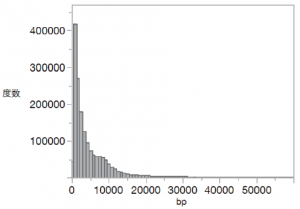 |
Read number | 1,670,827 | |
| Total read length | 7,655,872,327 | ||
| Average read length | 4,582 | ||
| Longest read | 566,198 | ||
| Shortest read length | 5 | ||
| N50 | 8,489(#268112) | ||
| N90 | 2,034(#956151) | ||

- こちらのアプリケーションノートのPDFダウンロード : こちら
- 製品情報詳細ページ: 自動DNA断片ゲル抽出システム BluePippin
- アプリケーションノート検索ページ(型番・キーワード・アプリケーションから検索可能)