日本ジェネティクスのアプリケーションノートとは?
- 当社製品を実際にご使用頂いた、正真正銘、日本国内の研究者様による評価データ
- 製品をご検討中の方はもちろん、すでにお使いのお客様におかれましても、類似の研究をされている他の研究者の方の事例集としてご活用頂けます
アプリケーション検索専用ページはこちら
メーカー名:日本ジェネティクス株式会社
下記のデータは、筑波大学 国際統合睡眠医科学研究機構 柳沢・船戸研究室 藤山 知之 様のご厚意により掲載させていただきました。
背景
睡眠は動物に普遍的に認められる行動ですが、これを制御する神経科学的メカニズムはいまだ不明です。睡眠・覚醒行動を制御するメカニズムや神経回路機能の解析等には、基礎研究による分子機構の解明が必要であり、現在はマウスを含む様々な動物種を用いた研究が進められています。 我々の研究室ではフォワード・ジェネティクスを用いて睡眠・覚醒の分子メカニズムを担う新規遺伝子の探索を行い、レム睡眠時間が顕著に短縮するマウス家系Dreamlessを樹立することに成功しました。さらに、その原因遺伝子が非選択的陽イオンチャネルNALCNをコードするNalcn遺伝子であることを見出しました(Funato, Fujiyama et al., Nature 2016)。
本アプリケーションノートでは、CRISPR/Casシステムを用いてN末端にFLAG-HAタグ配列を挿入したNalcn遺伝子改変マウス脳組織におけるNalcn遺伝子のmRNA発現量を調べるためにサンプルの調製を行いました。成体マウス大脳皮質細胞より本Kitを用いてmRNAを抽出後、逆転写によりcDNAを合成し、リアルタイムqPCR(qPCR)およびDropletデジタルPCR(ddPCR)によりNalcn遺伝子の発現量の解析を行いました。本アプリケーションノートではPCR実験までの一般的なサンプル調製のフローの中でFastGene製品を使用できた実例をご紹介します。
実験条件
今回、Nalcn遺伝子のタンパクコード領域のN末にFLAG-HAタグ配列を挿入した遺伝子改変マウス系統(NalcnFLAG-HA/+)3系統(mouse A, B,C)のマウス脳組織(大脳皮質)からRNAを抽出した。A, B, C の違いはCRISPRによって作成されたマウス系統の系統樹立に使用した最初の親マウスが異なる点である。
再現性をとるため、手法が異なるリアルタイムqPCRおよびDropletデジタルPCRにて、Nalcn遺伝子ターゲット領域1および2に対し、GAPDHをリファレンス遺伝子とした発現定量解析を行った。
1.RNA精製
| RNA精製キット | Q社キット | FastGene™ RNA Premium Kit |
|---|---|---|
| サンプル | マウス脳組織(大脳皮質)>20mg 各キット6個体ずつ使用 | |
| 溶解バッファー量 | 1,000μL | 600μL※ |
| DNase I 処理 | 未実施 | 推奨プロトコルどおり実施 |
| RNA溶出バッファー量 | 50μL | |
※ ラージインプット推奨量のとおり
2.逆転写反応
インプットRNA量 : 0.2μg
逆転写酵素名 : PrimeScript RT PCR Kit(#RR014A)(Takara)
〈反応組成反応プログラム〉
Total RNA 0.2 μg
Random 6mers(20μM) 0.5 μL
Oligo dT Primer(2.5μM) 1 μL
dNTPs(10mM each) 1 μL
RNase Free dH2O up to 10.5
(Total Volume 10.5 μL)
↓
アニーリング 65℃ ・ 5min
↓
5×Buffer 4 μL
RNase Inhibitor 0.5 μL
PrimeScript RTase 0.5 μL
RNase Free dH2O up to 20
(Total Volume 20 μL)
↓
酵素反応 42℃ ・ 30min
↓
変性 95℃ ・5min
3.リアルタイムqPCR
インプットcDNA量 : 希釈なし逆転写反応液 1μL
qPCR試薬 : SYBR® Premix Ex Taq™ II
(Tli RNaseH Plus)(#RR820A)(Takara)
qPCR装置 :ViiA™ 7リアルタイムPCRシステム
(Thermo Fisher Scientific)
4. デジタルPCR
インプットcDNA量 : 希釈なし逆転写反応液 1μL
ddPCR試薬 :ddPCR™ EvaGreen Supermix
(#1864034)(Bio-Rad)
dPCR装置 :QX200™ Droplet Digital™
PCR システム(Bio-Rad)
結果
結果1.マウス脳組織(大脳皮質)から精製したRNAの収量(ng/μL)
| Sample | Q社 RNA抽出kit | |
|---|---|---|
| mouse A | 1 | 273.0 |
| 2 | 305.5 | |
| 3 | 259.5 | |
| mouse B | 1 | 560.9 |
| 2 | 218.7 | |
| 3 | 340.5 | |
| 平均 | 326.6 | |
| SEM | 49.9 |
| Sample | FastGene™ RNA抽出kit | |
|---|---|---|
| mouse C | 1 | 259.2 |
| 2 | 171.6 | |
| 3 | 101.0 | |
| Wildtype | 1 | 259.9 |
| 2 | 332.2 | |
| 3 | 99.4 | |
| 平均 | 203.9 | |
| SEM | 38.8 |
各キットにそれぞれ6個体ずつ使用したため、単純に比較はできないが、Student’s t testでp値は0.08となり、n.s.判定であった。
両キットとも遜色なくRNA抽出ができているが、FGはQ社と比較して少し収量が低い、という印象だった。
これはサンプル間やDNase処理の有無などによる差と考察された。純度は申し分なかった。
補足)mouse A, B, CについてすべてNalcn遺伝子のタンパクコード領域のN末にFLAG-HAタグ配列を挿入した遺伝子改変マウス系統である。
A, B, C の違いは、それぞれ異なる親マウスを使用してCRISPRによるマウス系統の系統樹立がなされた点である。
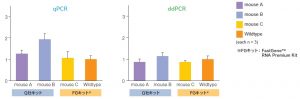
結果2.リアルタイムPCRとデジタルPCRによる定量
Wildtypeおよび同じgenotypeのNalcn遺伝子改変マウス(NalcnFLAG-HA/+)3系統(mouse A~C)のマウス脳組織(大脳皮質)から抽出したRNAを用い、リアルタイムqPCRおよびDropletデジタルPCRを実施した。
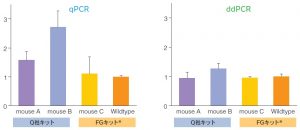
具体的には、Nalcn遺伝子ターゲット領域1および2に対し、GAPDHをリファレンス遺伝子とした定量解析を行った。今回、mouse A~Cの3系統とも、Nalcn遺伝子の発現制御に関わるプロモーター領域付近には変異を加えていないため、ターゲット遺伝子領域の発現量は全てwildtypeと同程度であることが期待された。
結果として、FastGene™ RNA Premium kitでRNA抽出したwildtypeとmouse Cでは、ターゲット遺伝子領域1、2とも、想定どおりほぼ同程度の発現量であることが確認された。
<Nalcn遺伝子ターゲット領域1>
<Nalcn遺伝子ターゲット領域2>

- こちらのアプリケーションノートのPDFダウンロード : こちら
- アプリケーションノート検索ページ(型番・キーワード・アプリケーションから検索可能)
- アプリケーションノート検索ページ(型番・キーワード・アプリケーションから検索可能)



















結果はこれまでのものと遜色なく、全く問題ない(むしろ良い)、との結論に至りました。
抽出したRNAの精製度が、これまでの手法で得られたものより非常に高かった点にも満足しております。
本来、NALCNチャネルは in vivo 脳組織内での検出が非常に困難なタンパクです。そこで、タグ配列を付加したNALCNタンパクを発現する脳組織に対し、タグ抗体を用いてより高精度にNALCNを検出することを目的とし、CRISPRで遺伝子改変マウスを作成しました。その結果、脳組織ライセートを用いたウエスタンブロットにて非常に特異性の高いシグナルを確認することができました。
今回のPCR実験結果より、野生型との比較でmRNA発現量には変化がないということが示され、NALCNタンパクそのものの発現量にもおそらく影響がないだろうことが期待されます。
今後は、タグ付加型タンパクを発現する遺伝子改変マウスを用いて、NALCNチャネルの分子特性についてより詳細に解析したいと考えています。