日本ジェネティクスのアプリケーションノートとは?
- 当社製品を実際にご使用頂いた、正真正銘、日本国内の研究者様による評価データ
- 製品をご検討中の方はもちろん、すでにお使いのお客様におかれましても、類似の研究をされている他の研究者の方の事例集としてご活用頂けます
アプリケーション検索専用ページはこちら
| アプリケーションノート 2018<02> 製品名:KAPA Hyper Prep Kit (KK8502, KK8503) メーカー名:KAPA BIOSYSTEMS 社 |
下記のデータは国立研究開発法人海洋研究開発機構 海洋生命理工学研究開発センター 生命機能研究グループ 平井 美穂様 高木 善弘様のご厚意により掲載させていただきました。
詳細な実験情報やデータは、下記の論文をご覧ください。
【発表論文】
雑誌名 : Microbes and Environments
タイトル : Library Construction from Subnanogram DNA for Pelagic Sea Water and Deep-Sea Sediments
著者 : Miho Hirai, Shinro Nishi, Miwako Tsuda, Michinari Sunamura,Yoshihiro Takaki,and Takuro Nunoura
Doi : 10.1264/jsme2.ME17132
URL : https://www.jstage.jst.go.jp/article/jsme2/32/4/32_ME17132/_article
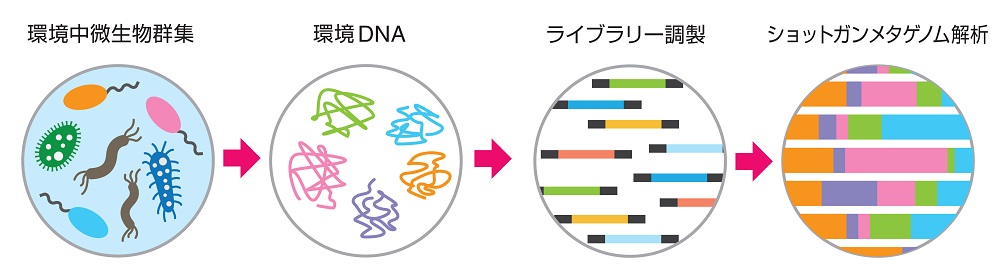
背景
深海水塊、海洋堆積物、海底下掘削試料から得られる環境DNA は、ショットガンメタゲノム解析を行う際に以下のような点が問題となります。
| 環境DNA のショットガンメタゲノム解析を行う際の問題点 | |
 |
• 採取機会・量が限定的である • 微生物マスが少ない • 環境 DNA中の夾雑物が多い |
|
• 1 ngに満たない極微量DNAからのライブラリー構築が必要となることがある |
|
そこで、ラボコンタミネーションの影響に注意しながら、超微量環境DNA1 pg からショットガンメタゲノム解析ライブラリー作成が可能かどうか試みました。
実験前に
微量のDNA 量を扱う場合、ラボ内でのコンタミネーションやチューブやチップへのDNA の付着を十分に留意する必要があります。
これらのリスクを回避するために、本事例で実際に行った注意点をご紹介します。
【注意点】
1)ラボでのコンタミネーションのリスク回避
・ 作業は全てテーブルコーチ(クリーンベンチ)内で実施する
・ オートクレーブ中は蒸気によるDNAの飛散が生じる等の、核酸が混入するリスクがあるため、使用するチューブのオートクレーブは避ける
・ DNA溶出には、購入後UV照射したdH2O(DNA Free Water)を使用する
・ フィルターチップを使用する
・ UV照射したクリーンルーム用のグローブを使用する
2)プラスチックへの吸着によるDNAロスのリスク回避
・ チューブは全て低吸着(low-Bind)タイプのチューブを使用する
・ ピペッティング回数は5回以内とする
(以前、上記の点を注意せずに実験を行った結果、コンタミネーション由来のデータが30%に達する事例がありました。)
ワークフロー
|
|||||||||||
| D N A 抽 出 |
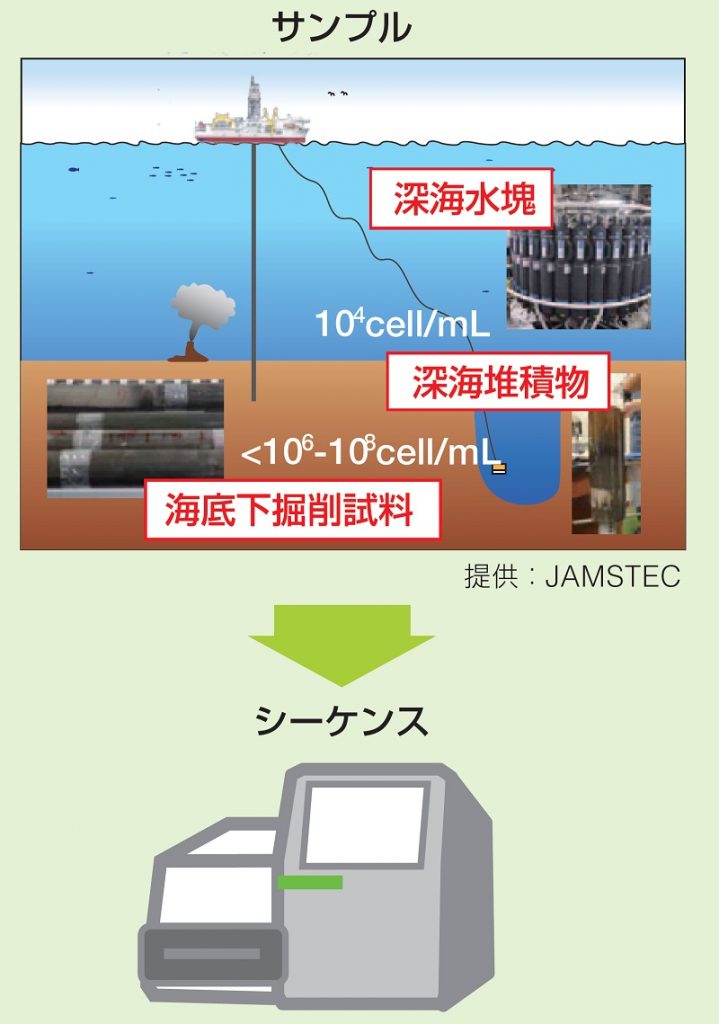
深海表層堆積物 〈シリカメンブレン法〉 PowerMax® Soil DNA Isolation Kit(MoBio Lab社) |
表層海水 〈粗抽出〉 ① 400 mM Tris-HCL, 60 mM EDTA, 150 mM NaCl,1% SDS(pH8.0) ② 3 M酢酸カリウム(pH4.8) ※試薬、バッファーはオーダーメード調製 ↓ 〈シリカメンブレン法〉 PowerSoil® DNA Isolation Kit (MoBio Lab社) |
|||||||||
 |
|||||||||||
|
糖や腐食物質などの環境DNA 中に混入してくる夾雑物
ライブラリー調製における各酵素反応 |
|||||||||||
| 反応前の夾雑物のクリーンナップが必要 | |||||||||||

| ラ イ ブ ラ リ Ⅰ 調 製 |
断片化 (物理的断片化/Covaris S220) |
KAPA Hyper Prep Kit |
|
|
|
インデックスアダプター濃度:150 nM 1 pg以上のライブラリー構築 標準プロトコール(1 ng以上が適用)では困難 ↓ 条件検討 インデックスアダプター濃度を150 nMにする ことで問題なくライブラリー構築ができた Amplification |
||
| 末端修復およびAテーリング | |||
| アダプターライゲーション | |||
| ビーズ精製 | |||
| ライブラリー増幅 | |||
| ビーズ精製 | |||
| シーケンス |
|||
|
MiSeq Reagent Kit v3(600 サイクル) |
 |
||
結果:ライブラリーの評価
詳細な実験情報やデータは、下記の論文をご覧ください。
【発表論文】
雑誌名 : Microbes and Environments
タイトル : Library Construction from Subnanogram DNA for Pelagic Sea Water and Deep-Sea Sediments
著者 : Miho Hirai, Shinro Nishi, Miwako Tsuda, Michinari Sunamura, Yoshihiro Takaki,and Takuro Nunoura
Doi : 10.1264/jsme2.ME17132
URL : https://www.jstage.jst.go.jp/article/jsme2/32/4/32_ME17132/_article
● クラスター解析
● SSU rRNA遺伝子群集構造による比較
深海表層堆積物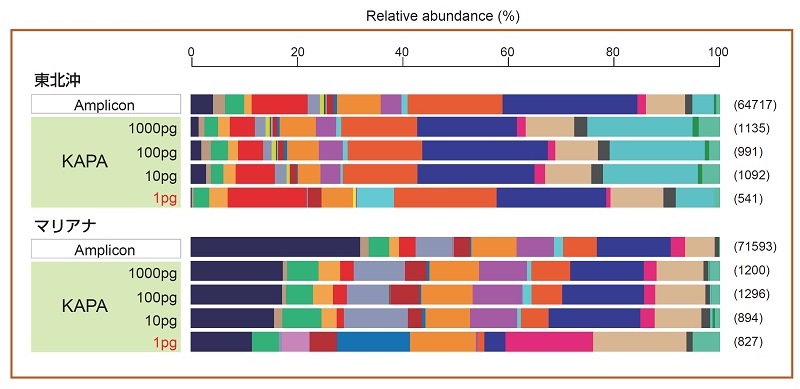 表層海水
表層海水
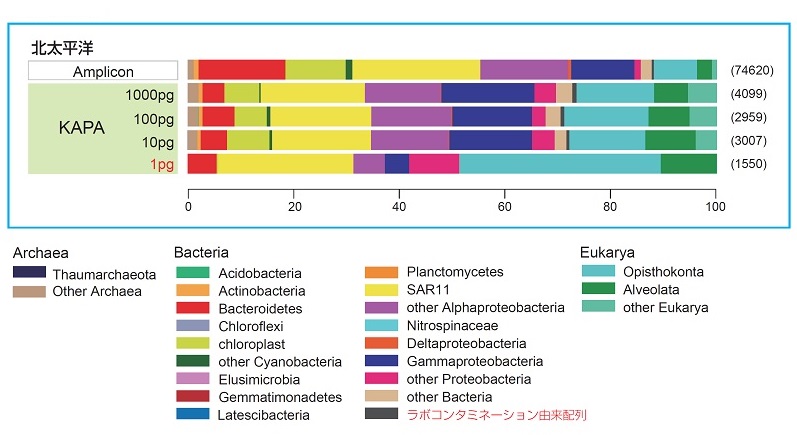
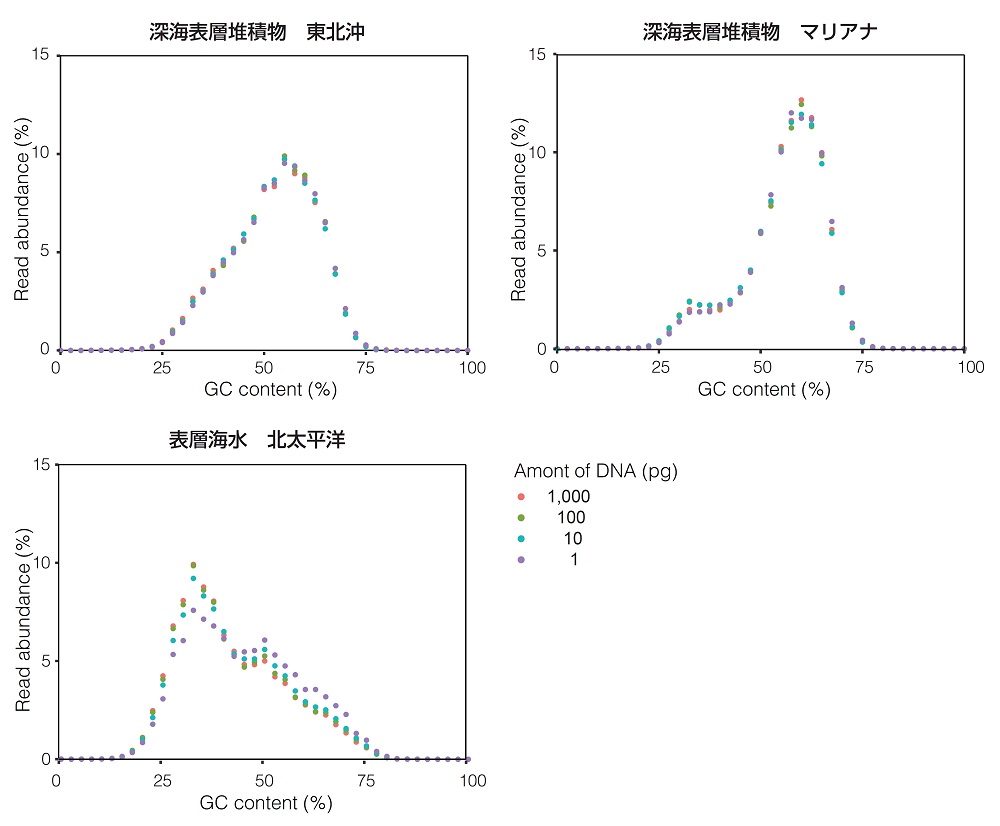
● リード毎のGC含量マップ
• 作業環境を改善することでコンタミネーションを抑えることができる
テーブルコーチ・水・試薬・道具などの作業環境を整えることで、1 pg でもラボコンタミネーションの影響を抑制できる

- こちらのアプリケーションノートのPDFダウンロード : こちら
- 製品情報詳細ページ: KAPA Hyper Prep Kit (for illumina)
- アプリケーションノート検索ページ(型番・キーワード・アプリケーションから検索可能)





















10 pg以上のDNAがあれば、アダプターの濃度とサイクルを変更することで、あとは全く同じプロトコルで、再現性よくライブラリーの構築が可能でしたので、幅広い分野に活用できるかと思います。
この実験系では、いかにラボコンタミネーションを軽減するか、DNAをなくさないかが重要ですので、是非その点に気をつけて研究を行っていただければと思います。