日本ジェネティクスのアプリケーションノートとは?
- 当社製品を実際にご使用頂いた、正真正銘、日本国内の研究者様による評価データ
- 製品をご検討中の方はもちろん、すでにお使いのお客様におかれましても、類似の研究をされている他の研究者の方の事例集としてご活用頂けます
- アプリケーション検索専用ページはこちら
| アプリケーションノート 2018<03> 製品名: KAPA Frag Kit(KK8600, KK8601, KK8602) メーカー名: KAPA BIOSYSTEMS 社 |
下記データは、国立研究開発法人 国立がん研究センター 研究所 エピゲノム解析分野 ユニット長 山下 聡先生の御厚意により掲載させていただきました。
はじめに
ライゲーションをベースとしたライブラリー調製の前処理として、シーケンスに供するDNAを、シークエンスに適した目的サイズに断片化することが求められます。
一方、サンプル数が多い場合、断片化条件を各サンプル毎に最適化し、同じサイズに断片化することは困難となります。
今回は、Ion ProtonシステムでのcDNAからのRNA-Seq用ライブラリー調製におけるcDNAの断片化処理において、DNA断片化用試薬 KAPA Frag Kitを用いたことで、サンプル間でバラつきが少なく、安定的に目的サイズのDNA断片が得られた事例をご紹介します。
ワークフロー
1. cDNAの精製
cDNA溶液2μL(cDNA 1μg相当)をAMPureXPで精製
10mM Tris HCl Buffer(pH8.0) 36μLで溶出し、35μLを回収
2.KAPA Frag Kit によるDNA断片化
① 氷上で酵素反応液の調製
AMPureXPで精製したDNA 35 μL
10X KAPA Frag Buffer 5 μL
KAPA Frag Enzyme 10 μL
—————————————————
Total 50 μL
② 断片化反応 37℃ 35min
*予備実験で決定した条件
③ 氷上でStop Solution 5 μLを添加し、ピペッティングで混合
3.AMPureXPによる精製
① ビーズ精製(×1.8)
反応液 55 μL
AMPureXP 99 μL
ピペッティングで十分撹拌後、5分静置し、マグネット上で上清を除去
② 洗浄
70%EtOH 洗浄 200μL x2回
③ 溶出
10mM Tris HCl Buffer(pH8.0) 36μLで溶出し、35μLを回収
4.PicoGreen®によるDNA定量およびTapeStationによるサイズ分布確認
5.ライブラリー調製
6.IonProtonによるシーケンス
結果
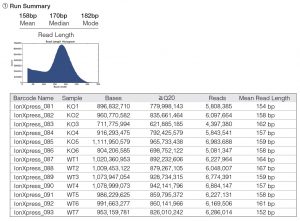
1.KAPA Frag Kit によるDNA断片化のサイズ分布
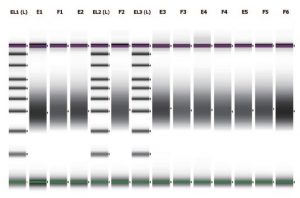
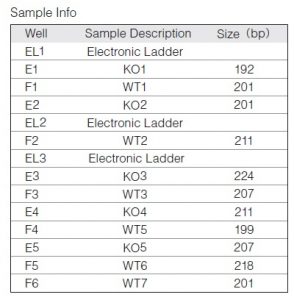
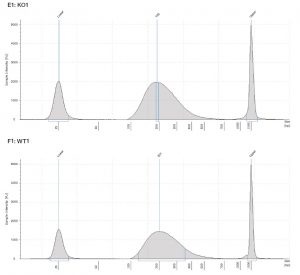
KAPA Frag Kit を用いた結果、均一なサイズ分布に断片化されていることが確認された。
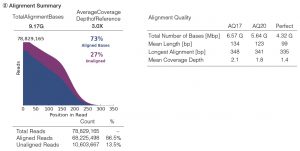
2.シーケンス結果
以下のように、期待どおりのシーケンス結果が得られた。
お客様のコメント
NGSライブラリのためのDNA断片化において、物理的な断片化方法も考えましたが、元のDNAの量やサイズがバラバラだと、この方法では常に同じサイズに揃えるのは難しい。
酵素を用いた容易な断片化法を求めていたところ、KAPA Frag Kitがあったので早速試しました。
元のDNAの長さが数キロだろうがゲノムだろうがほぼおなじ条件でよいようなので、条件検討は反応時間の確認だけで済みました。
あまりにも簡単に、均一なDNA断片化ができたので感動しました。
- こちらのアプリケーションノートのPDFダウンロード : こちら
- 製品情報詳細ページ: KAPA Frag Kit(KAPA HyperPlus Kitページ内)
- アプリケーションノート検索ページ(型番・キーワード・アプリケーションから検索可能)