日本ジェネティクスのアプリケーションノートとは?
- 当社製品を実際にご使用頂いた、正真正銘、日本国内の研究者様による評価データ
- 製品をご検討中の方はもちろん、すでにお使いのお客様におかれましても、類似の研究をされている他の研究者の方の事例集としてご活用頂けます
- アプリケーション検索専用ページはこちら
| アプリケーションノート 2018<05> 製品名: KAPA2G Fast HotStart PCR Kit(KK5500, KK5502) メーカー名: KAPA BIOSYSTEMS 社 |
下記データは、国立研究開発法人 国立国際医療研究センター研究所 ゲノム医科学プロジェクト
西田 奈央 様の御厚意により掲載させていただきました。
はじめに
DigiTag2 Assay(*1)は、PCR法をベースとし、超高速PCR反応用に開発された次世代DNAポリメラーゼ「KAPA2G Fast HotStart DNApolymerase」を用いることで、短時間で同時に96-plexや192-plexといった多検体のSNP 解析を可能とした画期的な手法(*2)です。
この手法は、ニワトリの系統分類(*3)や、肝炎や結核などのヒトの疾患ゲノム研究など、様々な実用性評価に応用されて来ました。
本アプリケーションノートでは、人為的に分解したDNAを用い、低品質・微量DNAを想定したDigiTag2 Assayによる民族識別(ethnic identification)の事例をご紹介します。
(*1)Nishida N, Mawatari Y, Sageshima M, Tokunaga K( 2012)Highly parallel and short-acting amplification with locus-specific primers to detect single nucleotide polymorphisms by the DigiTag2 assay. PLoS One 7(1):e29967.
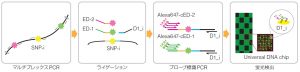
(*2) DigiTag2 Assayの特長(下図参照)
・数十から数百種のSNPを高い成功率で同時にタイピングすることが可能
・プライマー/ プローブを共通の設計水準でデザインするため、共通の実験条件でのSNPタイピングが可能(実験条件の検討が不要)
・解析対象によらず同一のDNAチップを用いたSNPタイピングが可能
・反応操作が簡便な上、一般的な実験装置(DNAチップスキャナー、サーマルサイクラー)でのSNPタイピングが可能
・10,000タイピング/ 日の処理能力
図:DigiTag2 assayの概要(96 plex SNP解析)
(*3)Shimogiri T, Nishida N, Kudo M, Niwa K, Nishibori M, Kinoshita K, Okamoto S, Maeda Y, Tokunaga K, Yasue H(2012)Genetic relationships between Japanese native and commercial breeds using 70 chicken autosomal SNP genotypes by the DigiTag2 assay. Animal Genetics 43(1)

方法
• DNAサンプル:ヒトゲノムDNA(HapMapサンプル, Coriell Institute)
*以下の3集団、各32 検体を使用した。
①①ヨーロッパ系アメリカ人(CEU:Utah residents with ancestry from northern and western Europe)
②②ナイジェリアのヨルバ族(YRI:Yoruba in Ibadan, Nigeria)
③③東アジア人(CHB:Han Chinese in Beijing & JPT:Japanese in Tokyo)
*段階的に分解したDNAを準備するため、各検体200ngのDNAを用い、反応条件(酵素濃度と反応時間)を振ってDNase Iで処理した後に、95℃ 5分で不活化した。
分解度合いを電気泳動で確認した後、25ngをDigiTag2 法によるタイピングに用いた。
• DigiTag2 Assay デザイン:
HapMap Phase II のすべてのSNPデータ(約400万種類)に基づき、CEU、YRI、CHB&JPTの3つの人類集団間でアリル頻度が大きく異なる32 種類のSNPを解析対象に選択した。
• Multiplex PCR条件
| PCR反応組成 | PCRプログラム | ||
| Template DNA | 25ng DNA | サーマルサイクラー | BioMetra T professional |
| Primer set | 25nM each | 95℃ 3min | |
| 5X KAPA2G Buffer | 1.5x(including 2.25mM Mg2+) | 95℃ 15sec | ×40 cycles |
| MgCl2 additional | 2.25mM Mg2+ | 68℃ 2min | |
| KAPA dNTP Mix | 0.2mM each | ||
| KAPA2G Fast HotStart DNA Polymerase | 0.04U | ||
| PCR-grade water | Up to 10 μL | ||
• Probe labeling PCR条件
| PCR反応組成 | PCRプログラム | ||
| Template DNA(ligation reaction solution) | 6μl | サーマルサイクラー | BioMetra T professional |
| labeled primer(Alexa555-cED-1 and Alexa647-cED-2) | 0.5μM each | 95℃ 1min | |
| D1 primer(D1_i) | 2.5nM each | 95℃ 15sec | ×30 cycles |
| 5X KAPA2G Buffer | 1.5x(including 2.25mM Mg2+) | 55℃ 120sec | |
| MgCl2 | additional 2.25mM Mg2+ | 72℃ 5sec | |
| KAPA dNTP Mix | 0.2mM each | ||
| KAPA2G Fast HotStart DNA Polymerase | 0.02U | ||
| PCR-grade water | Up to 12 μL | ||
• 蛍光検出およびSNP 解析
DNA チップスキャナー: GenePix 4000B
結果
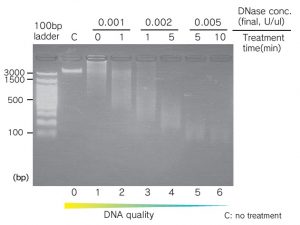
図1:DNaseI酵素処理によりDNAを人工的に断片化した。
酵素量と反応時間を調整することで段階的に断片化されている様子が分かる。
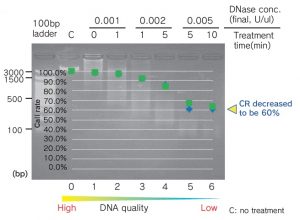
図2: 断片化DNAを用いてDigiTag2法による32SNPsタイピングを実施した。一番断片化が進んだDNAを使用するとCall rateは約60%まで低下した。
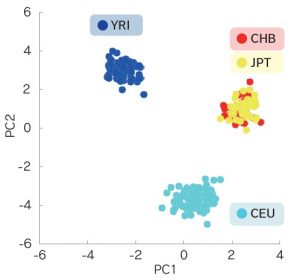
図3: HapMapサンプルを用いてDigiTag2法による32SNPsタイピングを実施した。
主成分分析(PCA)の結果、アジア系集団(CHB、JPT)、ヨーロッパ系集団(CEU)、アフリカ系集団(YRI)が3つのクラスターに分かれた。
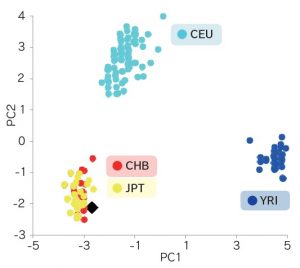
図4: 人工的に分解したHapMapサンプルを1例(JPTサンプル)加えてDigiTag2法による32SNPsタイピングを実施した。
20SNPsの遺伝子型を決定することができ、PCAの結果、アジア系集団のクラスターに入る様子が見えた。
お客様のコメント
96plexのマルチプレックスPCRでも一様な増幅が可能であることを確認しており、ヒト疾患ゲノム研究の推進に役立っています。
- こちらのアプリケーションノートのPDFダウンロード : こちら
- 製品情報詳細ページ: KAPA2G Fast HotStart PCR Kit
- アプリケーションノート検索ページ(型番・キーワード・アプリケーションから検索可能)