日本ジェネティクスのアプリケーションノートとは?
- 当社製品を実際にご使用頂いた、正真正銘、日本国内の研究者様による評価データ
- 製品をご検討中の方はもちろん、すでにお使いのお客様におかれましても、類似の研究をされている他の研究者の方の事例集としてご活用頂けます
アプリケーションノート検索専用ページはこちら
メーカー名:KAPA BIOSYSTEMS 社
以下のアプリケーションデータは埼玉県立がんセンター 腫瘍診断・予防科
山本 剛 様、 角田 美穂 様、 部長 赤木 究 様のご厚意により掲載させていただきました。
実験方法
遺伝性大腸がん原因遺伝子のひとつPMS2遺伝子をターゲットとした長鎖アンプリコンのシーケンスでは、特にATリッチ領域におけるライブラリー増幅バイアスによりリード数が減少し、カバレッジが低くなることが問題となっていた。
今回、現行法である「長鎖アンプリコンからNextera XTで作成したTagmentation後のライブラリー」において、KAPA Library Amplification Kit(KAPA HiFi HotStart ReadyMix)を用いてライブラリーを増幅することで、この問題点の改善を試みた。
ワークフロー
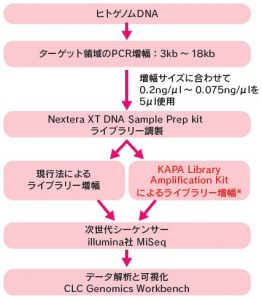
※KAPA Library Amplification Kitによるライブラリー増幅
① Tagmentationの中和ステップ後、25μlの反応液に2倍量(50μl)の AMPureXPを添加し、クリーンナップを実施(80%エタノール洗浄×2回)
② 15μl の10mM Tris-HCl, pH 8 あるいは PCR-grade water で溶出
③ 以下の条件でライブラリー増幅を実施
■反応組成
2×KAPA HiFi HS ReadyMix 25μL
Index 1 primer 5μL
Index 2 primer 5μL
Library DNA 15μL
50μL RXN
■PCR サイクル
initial Extension 72℃ 3min
Denaturation 98℃ 30sec
Denaturation 98℃ 10sec
Annealing 63℃ 30sec ×14cycles
Extension 72℃ 3min
Hold 10℃
<現行法>

PMS2遺伝子を標的とした長鎖アンプリコンシーケンスの結果
ATリッチ領域(例えば矢印の領域)などではリードが減少し、カバレッジの低下が見られる。
結果
KAPA Library Amplification(LA)Kitを用いてライブラリー増幅を行った結果、ATリッチ領域でのカバレッジの改善が見られ、より平均的なカバレッジを得ることができた。




他のサンプル(下段サンプルB)でも同様に、KAPA LA Kitを用いてライブラリー増幅した結果、カバレッジの改善が見られた。


- こちらのアプリケーションノートのPDFダウンロード : こちら
- アプリケーションノート検索ページ(型番・キーワード・アプリケーションから検索可能)