日本ジェネティクスのアプリケーションノートとは?
- 当社製品を実際にご使用頂いた、正真正銘、日本国内の研究者様による評価データ
- 製品をご検討中の方はもちろん、すでにお使いのお客様におかれましても、類似の研究をされている他の研究者の方の事例集としてご活用頂けます
アプリケーションノート検索専用ページはこちら
メーカー名:KAPA BIOSYSTEMS 社
下記のデータは 奈良先端科学技術大学院大学 バイオサイエンス研究科 細胞間情報学研究室
(高山研究室)久保健一様のご厚意により掲載させていただきました。
方法
ゲノムDNAテンプレートの調製
ゲノムDNAテンプレートの調製
① ペチュニア種子を、1/2濃度のMS塩を含む固形培地に無菌播種
② 播種後1週間でほぼ全ての芽生えの子葉が展開する。展開した子葉を1枚ずつ採集、8連PCRチューブに1枚ずつ集める。
③ DNA溶出バッファー(100mM Tris-HCl (pH8.0), 1M KCl, 10mM EDTA) 100μlを加え、ピペットの先端で子葉片を押しつぶす。
子葉組織から気泡を押し出す感覚で押しつぶし、組織をバラバラに壊さないことがコツ。
④ 95℃, 5 min インキュベートする。
⑤ 軽く遠心し、上清の一部をTEバッファーで10倍希釈する。
反応組成
⑤ の希釈DNA溶液 1.0μl
2×KAPA Plant Buffer 2.5μl
10μM Forward Primer 0.15μl
10μM Reverse Primer 0.15μl
KAPA3G Plant DNA Polymerase 0.05μl
100×KAPA Plant PCR enhancer 0.01μl
DW 1.14μl
Total 5.0μl※
※KAPA3G Plant PCR Kitのプロトコールでは、阻害物質の影響を軽減するため、反応量は50μlから検証を始めることを推奨しております。
結果に応じて反応量を減らすことが可能です。
プログラム
95℃ 5 min
95℃ 20 sec
54℃ 15 sec ×40サイクル
72℃ 30 sec
72℃ 2 min
解析
PCRプロダクト全量を用い電気泳動する。
結果
KAPA3G Plant PCRキットによるPCR結果
(対立遺伝子ABヘテロ接合体の次世代分離集団のジェノタイピングの一例)
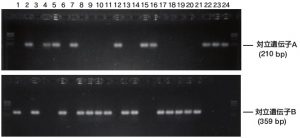
マーカー :φX174 HaeIII
lane 1-24 :芽生え子葉由来PCR産物
電気泳動条件 :1x TAEバッファー, 1.5 %アガロースゲル. 100V. 15 min. 反応液全量(5μl)をアプライ
対立遺伝子A(210bp)および対立遺伝子B(359bp)において、1 ~ 24の全てのサンプルでどちらか一方のバンドのみ増幅がみられた。
結果として、KAPA3G Plant PCRキットでは、各個体で安定して特異的に増幅が見られ、対立遺伝子を持つ個体と持たない個体の区別がはっきりでき、遺伝子型が容易に特定できた。

- こちらのアプリケーションノートのPDFダウンロード : こちら
- アプリケーションノート検索ページ(型番・キーワード・アプリケーションから検索可能)




















複数遺伝子の遺伝的連鎖を観察するため、多検体に対して複数のプライマーセットで増幅を行い、安定な結果を得る必要があり、そのためのテンプレート調製方法や反応ボリュームの最適化を行った。本法で作製したテンプレートを用い、1 ヶ月以上安定な増幅が可能であったので、多数の遺伝子の連鎖を解析することができた。
これまで他社キットでは安定に増幅しなかったが、KAPA3G Plant PCR キットでは、プライマーペアの間で増幅にばらつきが小さく、安定していて使いやすい。