日本ジェネティクスのアプリケーションノートとは?
- 当社製品を実際にご使用頂いた、正真正銘、日本国内の研究者様による評価データ
- 製品をご検討中の方はもちろん、すでにお使いのお客様におかれましても、類似の研究をされている他の研究者の方の事例集としてご活用頂けます
アプリケーションノート検索専用ページはこちら
メーカー名:KAPA BIOSYSTEMS 社
以下のアプリケーションデータは理化学研究所情報基盤センター
バイオインフォマティクス研究開発ユニット 笹川洋平 様のご厚意により掲載させていただきました。
はじめに
KAPA Hyper Prep Kitは1ngまでのdsDNA(1ng ~ 1μg)からのライブラリー調製に適したキットですが、今回は、更に微量な(10-1,000pg)に対応できるようプロトコールの最適化を実施し、運用している事例(LIMprep2)をご紹介いたします。
図1)インプットDNAに対するラブラリーへの変換効率
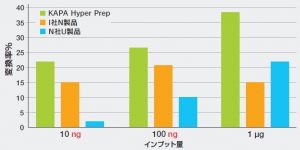
KAPA Hyper Prep KitではインプットDNAを、より多くのアダプター付加済みライブラリーに変換させます。
コバリスで断片化されたDNAから作成されたライブラリーは、アダプター・ライゲーション後に KAPALibrary Quantification Kitにより定量されました(上図参照)。
KAPA Hyper Prep KitはDNAインプット量(10ng、100ng、1μg)に関わらず、最も高いアダプター付加済みライブラリーへの変換率を示し、より少ないサイクル数での増幅で1μgのライブラリーを作製することが出来ました。(KAPA Biosystems社データ)
LIMprep2 Workflow
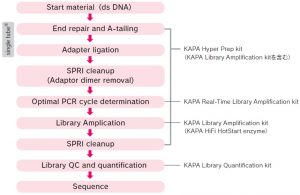
※これらのステップはひとつのチューブ内で実施

磁気ビーズによる精製ステップには、微量サンプル用マグネットスタンド
MagnaStand YS-Model(8連×0.2mlPCRチューブ用Cat#FG-SSMAG2)を使用
http://bit.accc.riken.jp/protocols/
結果
事例データ①
1ng fragmented 200bp genomic DNAを用い、アダプター濃度の最適化を実施した。
図2)アダプター濃度とライブラリー転換効率の検証
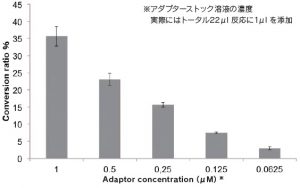
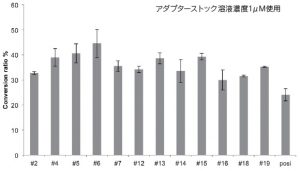
図3 使用したindexの異なるアダプター間の比較
事例データ②
Quartz-seq※のcDNAからテクニカルレプリケートとして、LIMprep2プロトコールを用いて複数回ライブラリーを作製し、MiSeqでシーケンスした。
その結果、概ね1-2M readsのデータを得られ、少ないリードでも高い相関性を示し、Quartz cDNAから高い精度でシーケンスが実施可能であることを確認した。
発現定量し、散布図で確認した。
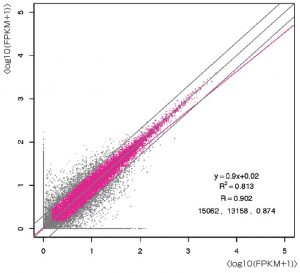
少ないリード数でも十分な相関が得られている
Adaptorを除いて、FASTQCを実施した結果データ
■Per base sequence quality
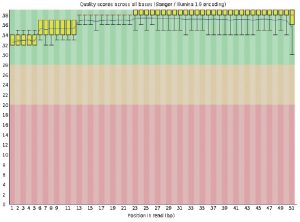
リード50bpでも高いクオリティを維持
■Per sequence quality scores
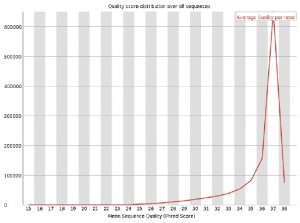
クオリティスコアは37が多い
■Per base sequence content
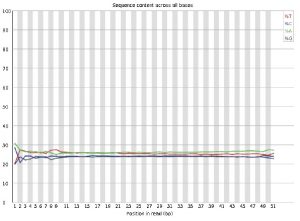
読み初めから塩基に偏りがない
■Per base GC content
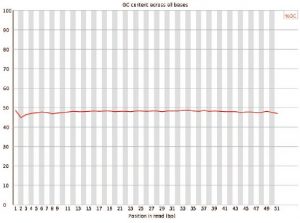
50bpまでGC含有率も偏りがない
Adaptorを除いて、FASTQCを実施した結果データ
■Per sequence GC content
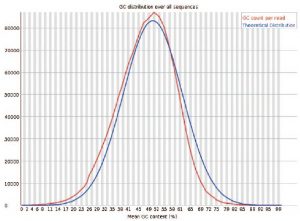
理論値と同様のリードGC含有率の正規分布
■Per base N content
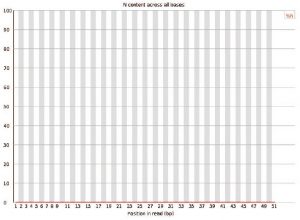
Nは現れなかった
■Sequence Length Distribution
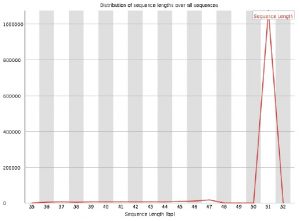
リード長は51bpとなった
■Sequence Duplication
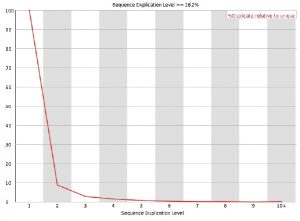
重複リードはほとんどなかった

- こちらのアプリケーションノートのPDFダウンロード : こちら
- アプリケーションノート検索ページ(型番・キーワード・アプリケーションから検索可能)



















KAPA Hyper Prep kitは、工程の単純化と精度の担保と向上を同時に達成した良いキットだと思います。
KAPA社は、安全マージンを多めにとってプロトコルを出しているため、1ng以下の微量におけるデータがありませんでしたが、我々が確かめたところ微量領域においても高いライブラリー変換効率を保つことが確かめられました。最適化プロトコルは我々のラボWebサイトからダウンロードできます。
少ない工程で、液を上から足していけば達成できるので、大多数のユーザーにとってファーストチョイスになると思いました。