日本ジェネティクスのテクニカルノートとは?
発売したばかりの新製品の性能評価や、既存製品の最適な条件を追求するための条件検討をすることでお客様に心からご納得頂いた上で商品をお使い頂けるよう、様々な評価試験を行っています。
採用前の検討資料として、または採用後の最適条件検討資料としてご活用ください。
また、当データに関するご質問はお気軽にページ下部のフォームよりお問い合わせください。
概要
ゲル電気泳動を用いたDNA量の定量方法は、あまり定量的ではないという印象が強く、「“半”定量」などと言われている。本テクニカルノートでは、実際、どの程度の定量性が得られるのか、Midori Green XtraとFAS-Digi PROを用いたゲル電気泳動像をImageJで画像解析を行い、定量域の大きさ(ダイナミックレンジ)と検出分解能を評価した。
評価方法
実験① ダイナミックレンジの検証
実験② 検出分解能の検証
実験①:ダイナミックレンジの検証
方法
ダイナミックレンジを評価するためにDNAラダーを段階的に量を減らして、2%アガロースゲルにアプライし、1,500 bp のバンド輝度をImageJを用いて評価した。
実験①結果 電気泳動
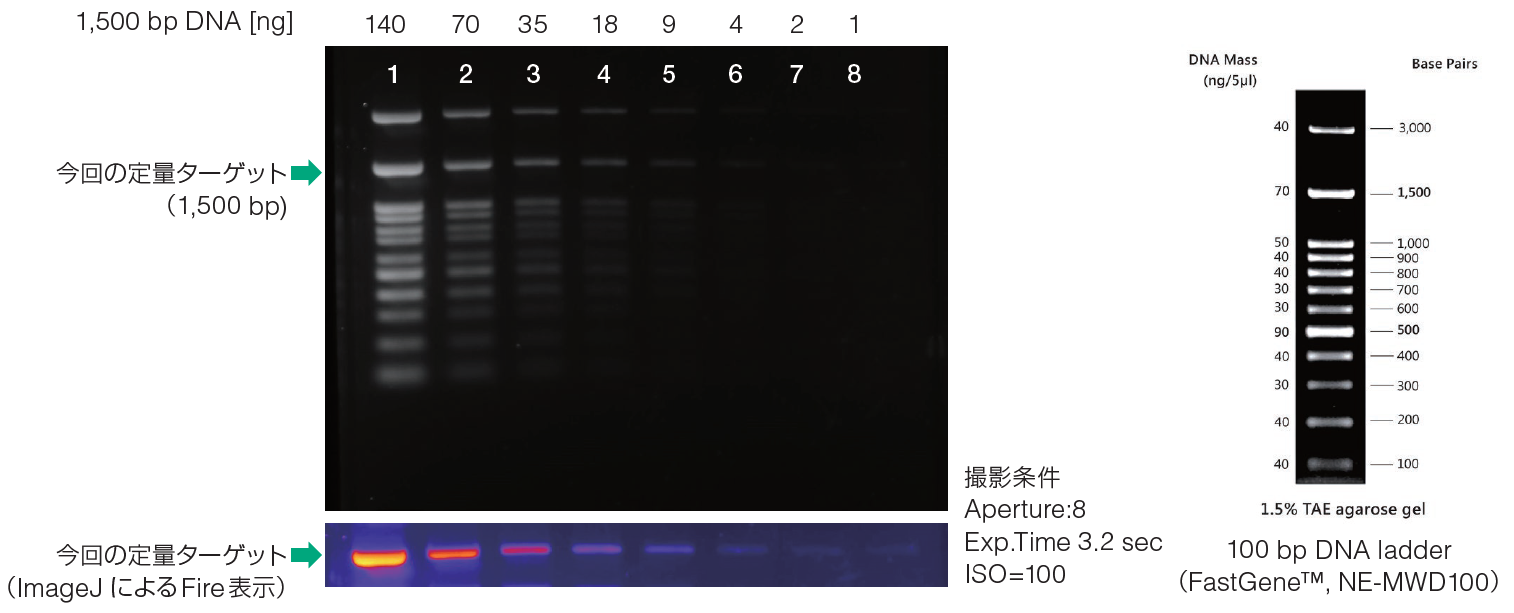
実験①結果 相対バンド輝度
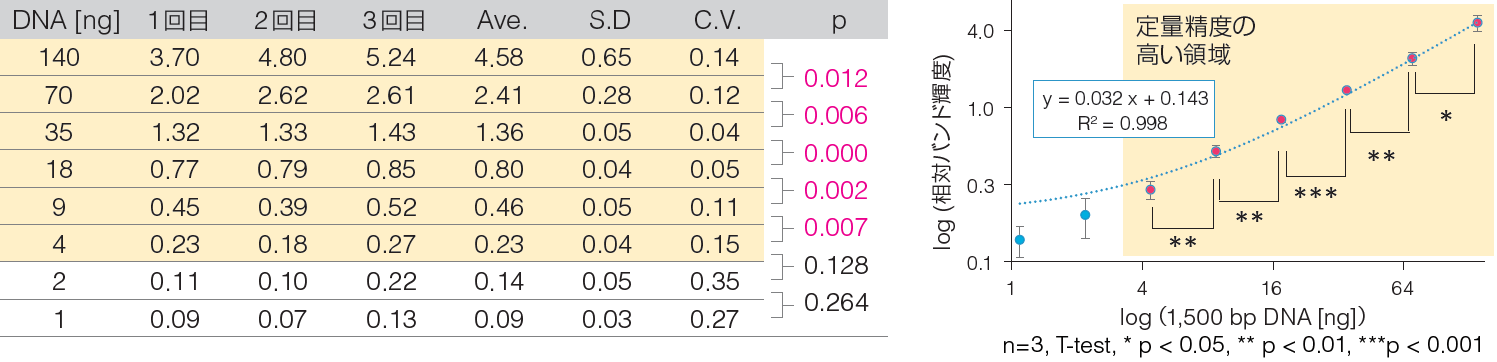
- 1500 bp DNA、140 ng ~ 1 ng の間で、R2=0.998の精度で線形性が得られた。
- 1500 bp DNA、140 ng ~ 4 ng の間で、倍量のDNAを、n=3の施行で有意な差をもって検出できる精度があった。
→定量可能域は140 ng ~ 1 ngであるが、特に定量精度が高い領域は140 ng ~ 4 ngである。
バックグラウンドの影響を受けやすい条件下(DNA量が少ないとき)では、定量精度が下がり、直線性が得られなくなっている。このことは、ダイナミックレンジの広い、精度の高い定量を実現するためには、可能な限り低バックグラウンドとなるよう気を配る必要があることを意味している。
結論①
アガロースゲル電気泳動は、35倍量程度の差のDNA量を扱うことができるダイナミックレンジを持つことが分かった。
実験②:検出分解能の検証
方法
検出分解能を評価するために、DNA量 18 ng ±10%, 20%, 50%の範囲で変化させ、 18 ng のバンド輝度を比較した。
実験②結果 電気泳動
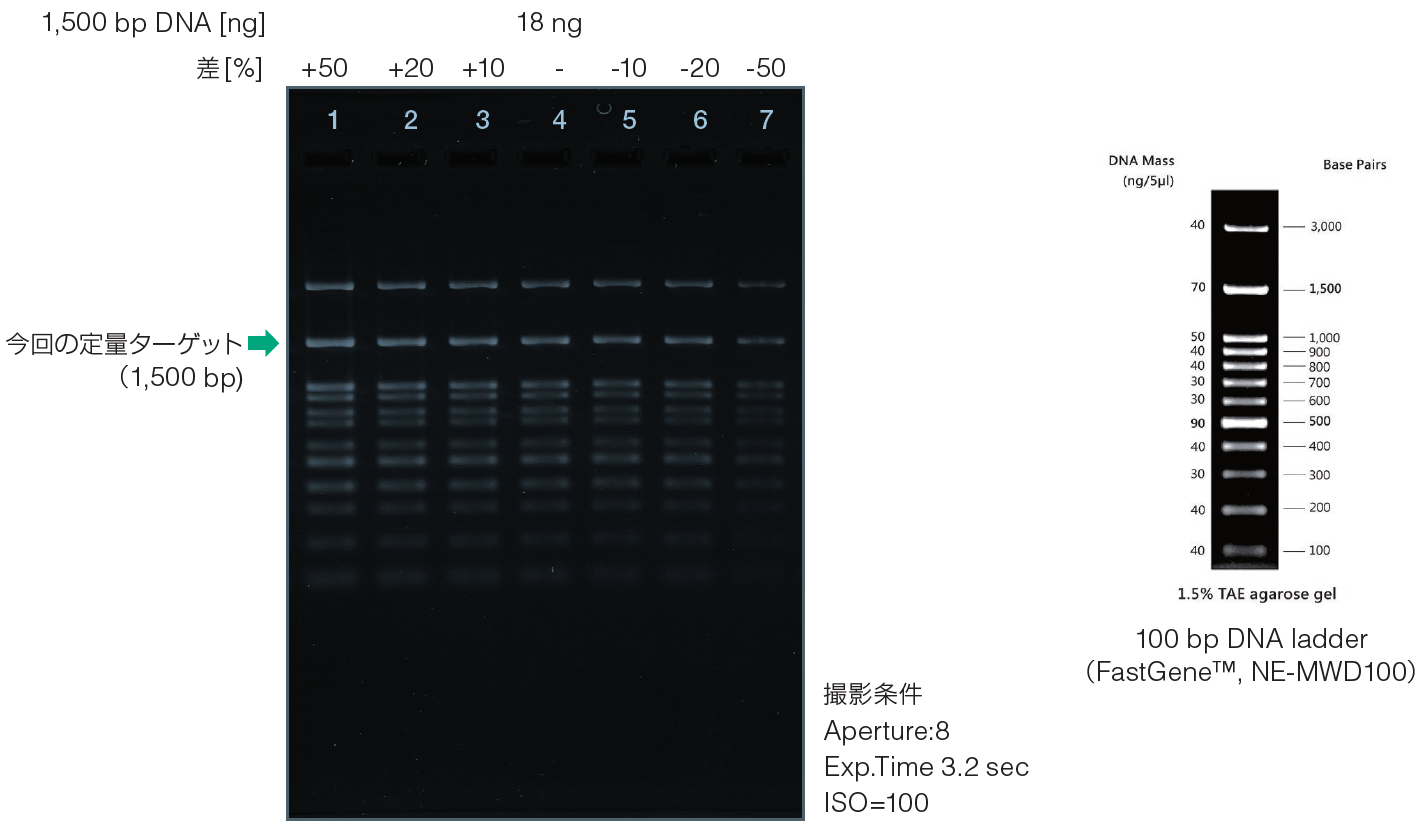
実験②結果 相対バンド輝度
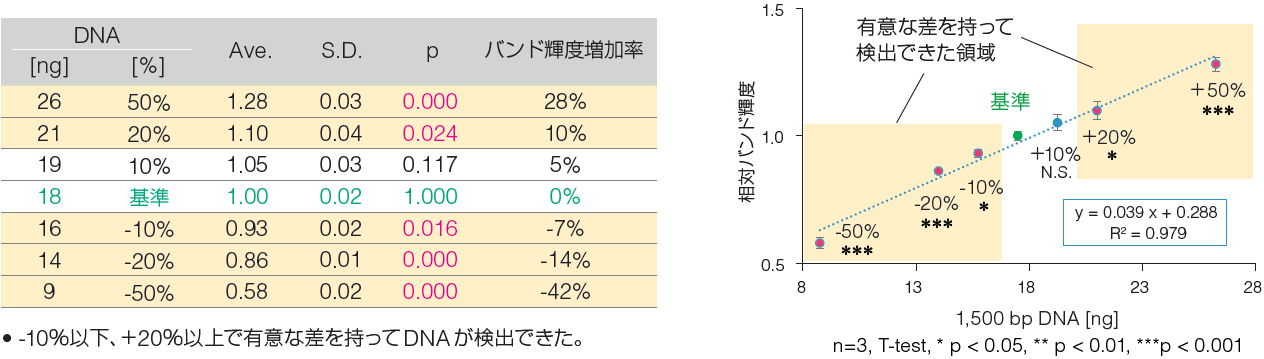
n=3の施行で、少なくとも20%の差を有意な差を持って検出分解能を有することが分かった。しかしながら、DNA量を50%に増やした際に、バンド輝度増加率は28%にしか至らなかった。これは、定量値は、依然、バックグラウンドの影響を含有しており、絶対量を反映しているわけではないことを意味している。
このように、一般的に、電気泳動の定量評価は、ゲル由来のバックグラウンドの影響を受けやすい。このためゲル電気泳動を用いた定量法では、既知のDNA量と比較するなど、必ず指標を置き、相対的に評価する必要があることが理解できる。
結論 ②
アガロースゲル電気泳動の検出分解能は、20%程度の差は有意差を持って検出できるポテンシャルを有する。
- 本テクニカルノートの結果は、ゲル電気泳動の定量性を示す1つの指標である。しかし実際には、手技、装置に依存する部分も多く、本方法以外のゲル電気泳動に関する定量性は言及できない。したがって、あくまでも、Midori Green XtraとFAS-Digi PROを用いたゲル電気泳動の結果である点には注意が必要である。
- 一般的に、リアルタイムPCRを用いた定量でも検出分解能は10%~ 20%程度と考えられている。
このことを踏まえると、Midori Green XtraとFAS-Digi PROを用いたゲル電気泳動による定量は、定量条件を吟味することによって、
リアルタイムPCRと遜色ない検出分解能を有することを意味している。
この点を加味すると、本定量に用いたMidori Green XtraやFAS-Digi PROは、定量に適した試薬とゲル撮影装置であると言える。
実験方法
<試薬・機器>
泳動用ゲル(2%アガロースゲル)
試薬:アガロース(FastGene™, Cat.No. NE-AG02)
溶媒:1× TAE バッファー (nacalai tesque, Cat.No.35430-61)
DNAサンプル
100 bp DNA ladder(FastGene™, Cat.No. NE-MWD100)
電気泳動装置
SafeBlue Electrophoresis System(Major Science, Cat.No. MBE-150-Plus)
泳動バッファー
1×TAE バッファー
核酸染色試薬
Midori Green Xtra(FastGene™, Cat.No. NE-MG10)
ゲル撮影装置
FAS-Digi PRO(日本ジェネティクス株式会社, Cat.No. GP-07LED/PC)
<方法>
- 2% アガロースゲルにDNAサンプルをアプライし、100 V、35分間、電気泳動を行った。
- 泳動後、Midori Green Xtra 10 μL / TAE 100 mLの溶液を用いて、30分間浸透、染色を行った。
- FAS-Digi PROを用いて画像を取得した。
- 画像をImageJを用いて解析した。
Supporting Information:ImageJを使用したバンド輝度定量方法
① ゲルの撮影
Aperture、Exp. Time、Sensitivity 撮影条件を指定して撮影する。
(ファイルの保存形式は tiff を選択する。)
※複数の画像を比較する場合は、比較する画像間でこの値は一定でなければならない。
※ここで、定量性が担保される明るすぎず、暗すぎない画像を取得することがポイント。画像取得条件がわからない場合は、数条件のExp.Timeで画像取得する。
② ImageJに画像を取り込む
③ Split Channelsで8 bit 単色画像に変換する。
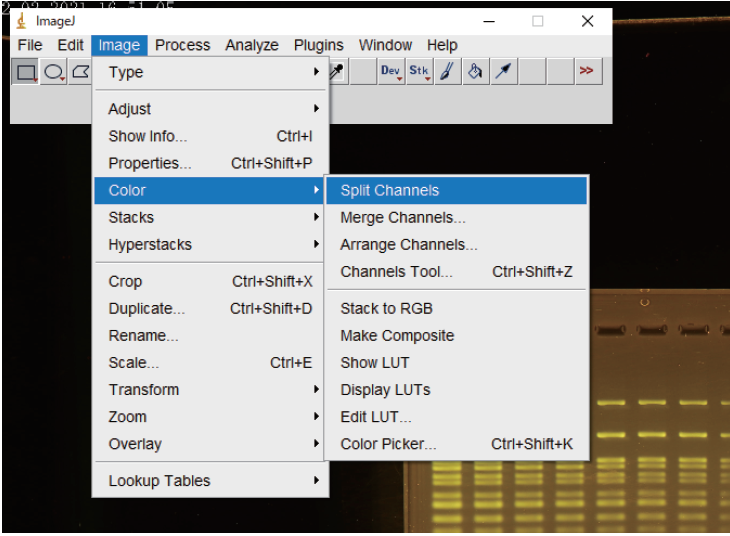
FAS-Digi PROで得られる画像はRBG(赤、青、緑)3色8 bit のTIFF画像である。ここの作業で1ピクセルの輝度を単色256段階で評価できるようになる。
④ 3分割された画像の緑(green)の色調を検出した画像を選択する。
青(Blue)、赤(Red)と記載のあるファイルは今回は定量に用いない。
⊠を押して消してよい。
※ここで選択する画像は、必ず緑である必要はないが、画像間で比較する際は、必ず同色で比較すること。
⑤ HiLoを選択し、バンドが赤表示されていないことを確認する。
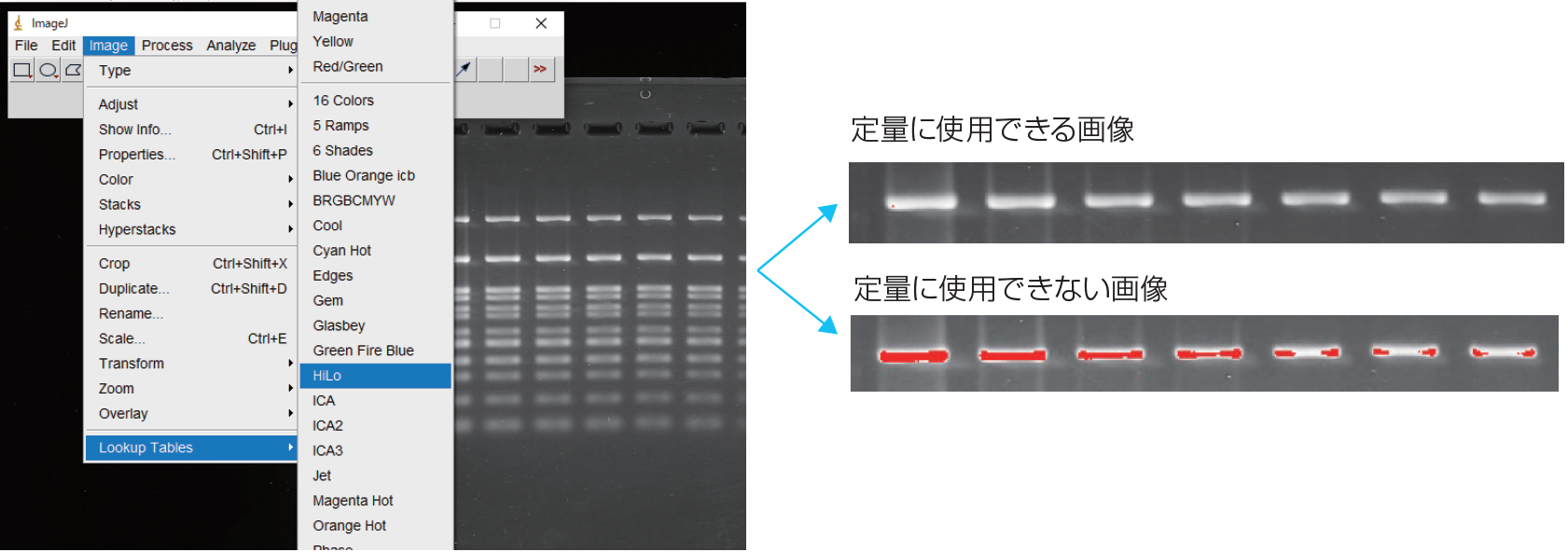
HiLoは画像の各ピクセルが256段階の1を青、256を赤で表示する機能である。
2 ~ 255と異なり、256は“256以上”のすべての輝度を含むため、定量的に評価できていないピクセルを意味する。
このため、定量に使用する領域が定量範囲内に収まっているかここで確認する。
※赤で表示されてしまう場合は、露光時間(Exp.time)を短くして、ゲル画像を取得しなおす。
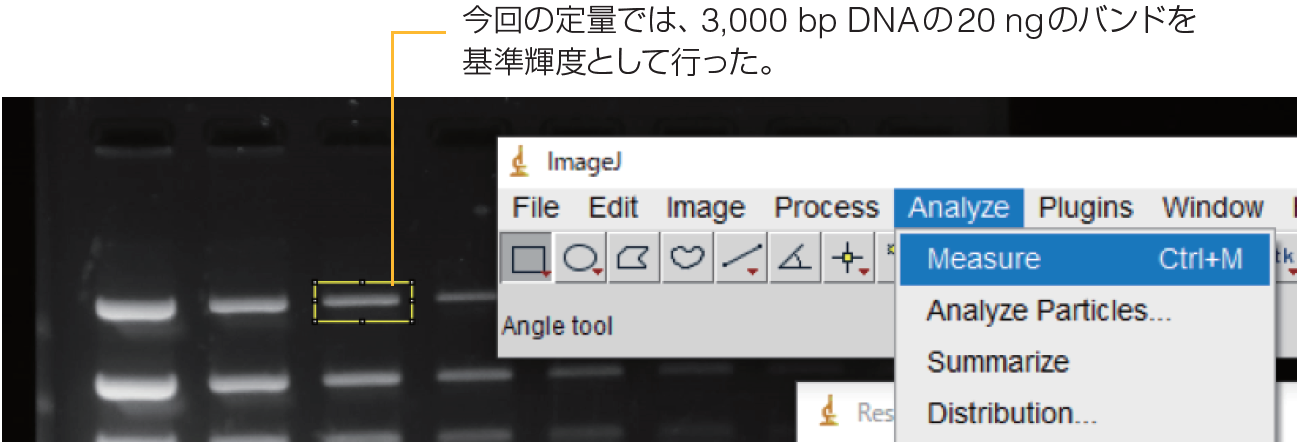
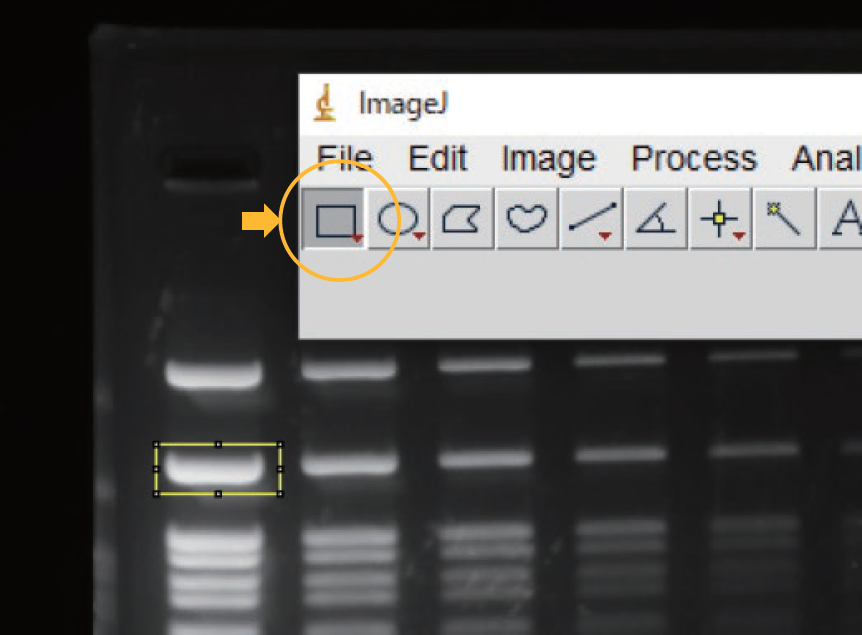
⑥選択モード四角を選択し、バンド全体を四角で囲い、定量するエリアを指定する。
⑦ Measureを選択し、バンドの輝度を測定する。
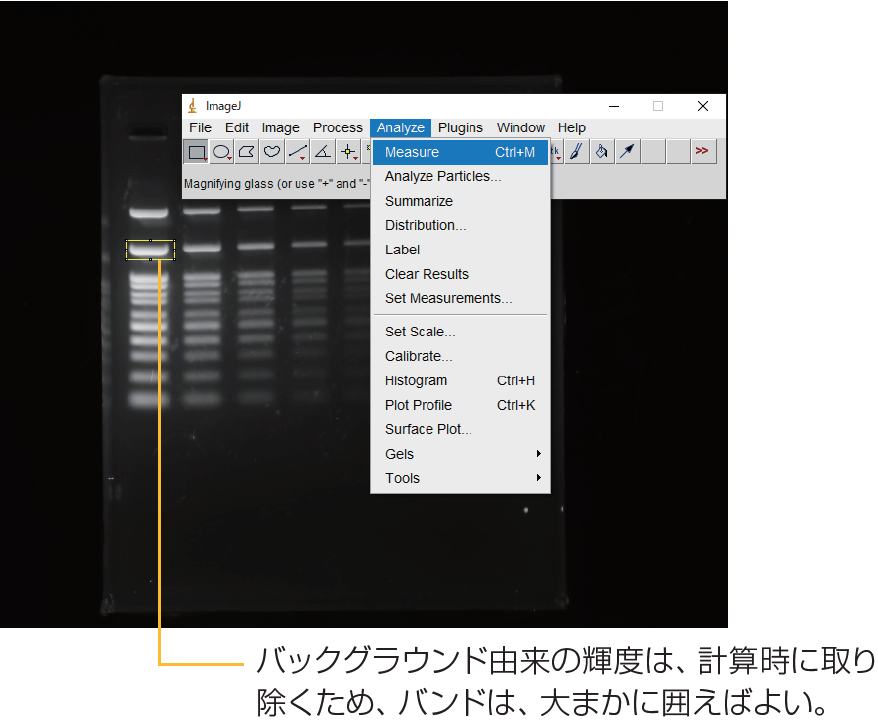
⑧ 同様の方法で、バンドの無いエリアを指定し、バックグラウンドの測定を行う。
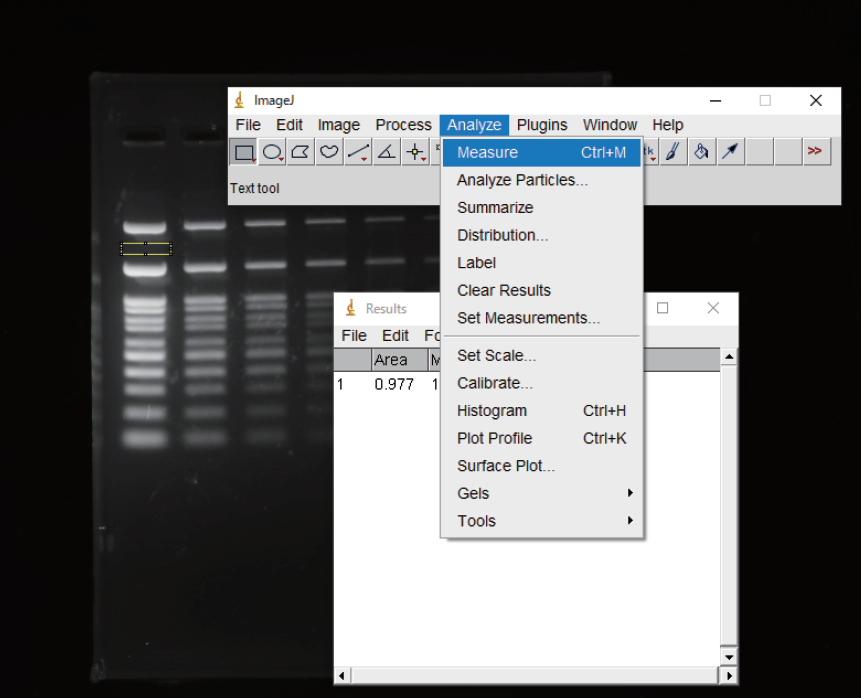
⑨ 定量したいバンドそれぞれについて④、⑤の工程を繰り返す。
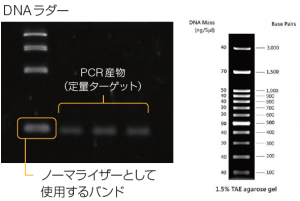
⑩ノーマライザーの輝度を測定する。
ノーマライザー:全体的な輝度を補正するための基準となるバンド
ゲル毎に既知の量のDNAを泳動し、これを基準輝度として、ゲル間で生じた輝度の補正を行う。
電気泳動の際に、いつも同じ量のDNAラダーを合わせて泳動するだけで、定量する際の補正に用いることができる。
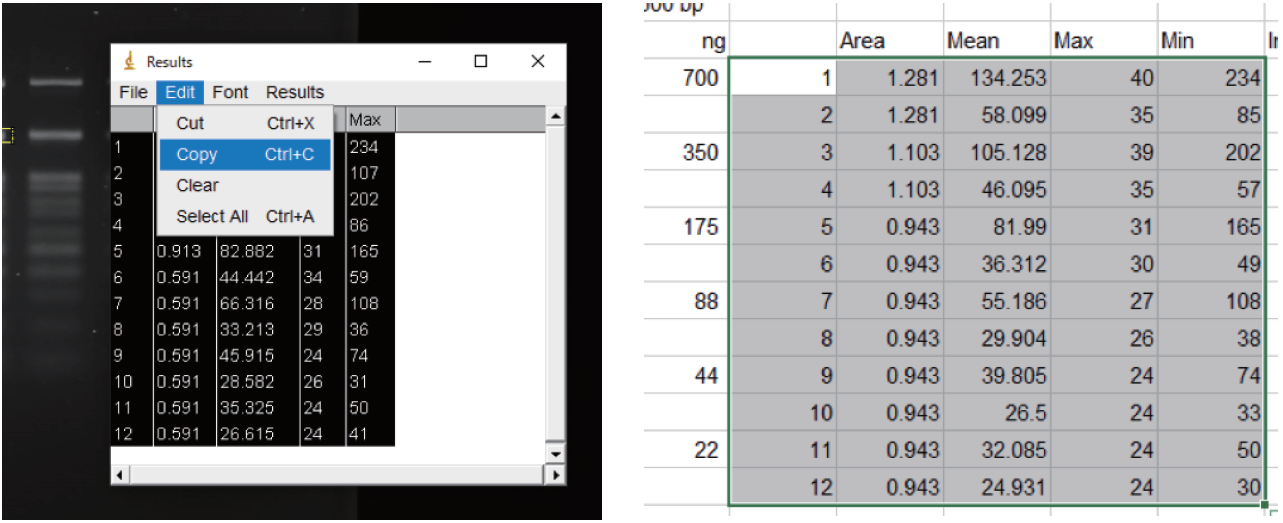
⑪ 測定結果をコピーし、エクセルにデータを張り付ける。
⑫ 各バンドの輝度を計算する。
計算に使用する値は以下の3つ
[a]:定量ターゲットのMean(四角で囲ったバンドエリアの平均輝度)
[b]:定量ターゲットのArea(四角で囲ったバンドエリアの面積)
[c]:バックグラウンドのMean(バックグラウンドとして囲ったエリアの平均輝度)
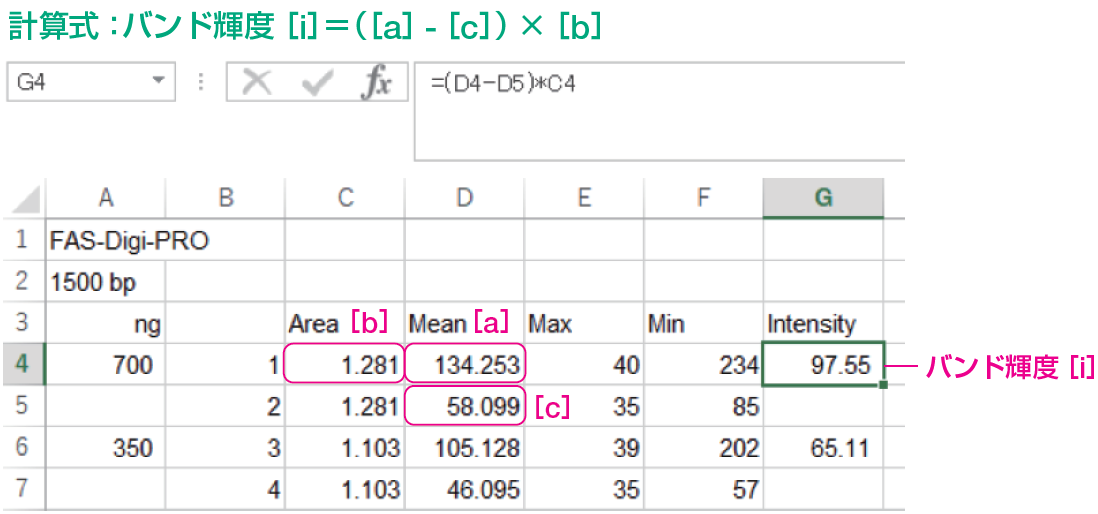
⑬ ノーマライザーのバンド輝度でゲル間の輝度の補正を行う。
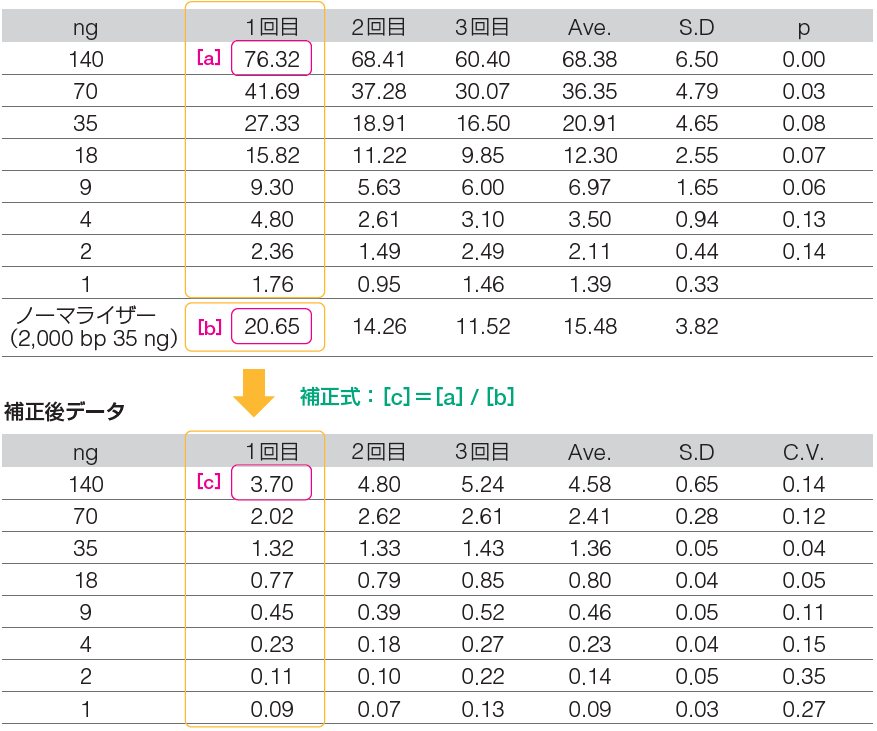
⑭ 試行回数分の輝度補正を行い、その平均値を算出する。
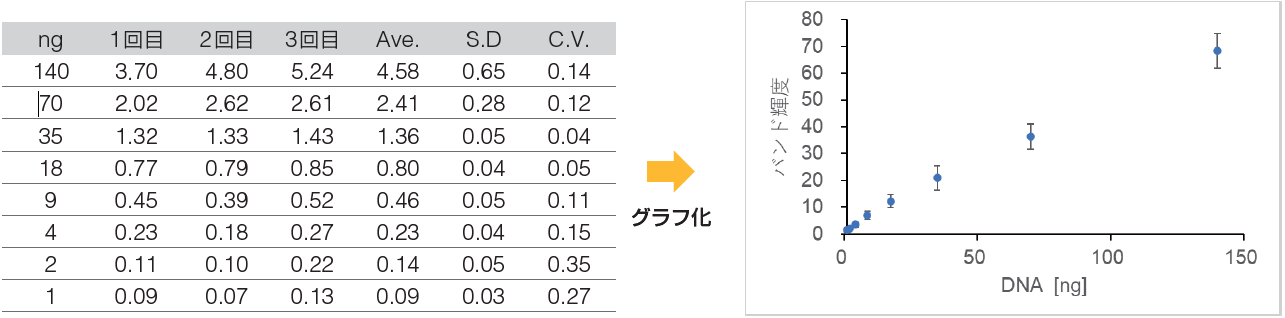
※本測定方法について
ImageJには各泳動レーンの泳動距離に応じた輝度を測定する、ゲル解析用の機能が備わっているが、データの扱い方や操作が煩雑である。
このため、本テクニカルノートでは、既述の測定方法を採用し解析を行った。