日本ジェネティクスのアプリケーションノートとは?
- 当社製品を実際にご使用頂いた、正真正銘、日本国内の研究者様による評価データ
- 製品をご検討中の方はもちろん、すでにお使いのお客様におかれましても、類似の研究をされている他の研究者の方の事例集としてご活用頂けます
アプリケーション検索専用ページはこちら
| アプリケーションノート 2018<12> 製品名:FastGene™ RNA Premium Kit(FG-81006, FG-81050, FG-81250) FastGene™ Scriptase Basic cDNA Synthesis(NE-LS62) FastGene™ Scriptase Ⅱ cDNA Synthesis(NE-LS63) メーカー名:日本ジェネティクス株式会社 |
下記のデータは、東京大学 大学院理学系研究科 生物科学専攻 古賀皓之 様のご厚意により掲載させて頂きました。
背景・目的
私は植物の多様な葉の形態の進化メカニズムを、発生の観点から理解するための研究をおこなっています。
私の所属する研究室では、葉の発生の仕組みを調べるためにさまざまな非モデル植物をつかって研究を行なっていますが、私は特に水辺に育つ植物、いわゆる水草、を主な実験材料としています。
今回、これら水草の一種であるオオバコ科のミズハコベを用いて、RNA抽出法の検討と、逆転写酵素反応の比較検討を行いました。
FastGene™ RNA Premium Kitを使ったRNA抽出では、操作の一部を変更することで、十分な量のRNAを抽出することに成功しました。また、逆転写酵素反応では、高発現の遺伝子だけでなく、発現量の低い目的転写産物と長鎖の受容体遺伝子で転写が行なえることを確認できました。
私の所属する研究室では、葉の発生の仕組みを調べるためにさまざまな非モデル植物をつかって研究を行なっていますが、私は特に水辺に育つ植物、いわゆる水草、を主な実験材料としています。
今回、これら水草の一種であるオオバコ科のミズハコベを用いて、RNA抽出法の検討と、逆転写酵素反応の比較検討を行いました。
FastGene™ RNA Premium Kitを使ったRNA抽出では、操作の一部を変更することで、十分な量のRNAを抽出することに成功しました。また、逆転写酵素反応では、高発現の遺伝子だけでなく、発現量の低い目的転写産物と長鎖の受容体遺伝子で転写が行なえることを確認できました。
実験方法
RNA抽出
| サンプル: オオバコ科水草 Callitriche palustris のシュート頂 サンプル量は7.4~10.9mg(RNA 抽出・結果の項参照) 破砕方法: 50 μL のLysis buffer(buffer RL またはbuffer RLT)中で 凍結した組織をジルコニアビーズで破砕 |
 |
 |
RNA抽出キット
■ 1-1. Q社(植物RNA抽出キット)- オリジナルプロトコルに準拠
■ 1-2. FastGene™ RNA Premium Kit(original large-input protocol)
■ 1-3. FastGene™ RNA Premium Kit(modified large-input protocol)
■ 1-1. Q社(植物RNA抽出キット)- オリジナルプロトコルに準拠
■ 1-2. FastGene™ RNA Premium Kit(original large-input protocol)
■ 1-3. FastGene™ RNA Premium Kit(modified large-input protocol)
① 抽出後、NanoDrop™とQubit™(Thermo Scientific)による定量を行なった。
② 抽出したtotal RNA の一部を用いてBioanalyzer-Agilent RNA 6000 nano kit(Agilent Techonologies)による品質チェックを行なった。
② 抽出したtotal RNA の一部を用いてBioanalyzer-Agilent RNA 6000 nano kit(Agilent Techonologies)による品質チェックを行なった。
逆転写酵素反応
RNA は 1-3. FastGene™ RNA Premium Kit(modified large-input protocol)で抽出した2 サンプルから total RNA 500 ng( Qubitでの測定した収量)を使用した。
逆転写酵素反応キット
■ 2-1. FastGene™ Scriptase Basic cDNA Synthesis
■ 2-2. FastGene™ Scriptase Ⅱ cDNA Synthesis
■ 2-3. I 社
■ 2-4. T社
すべてのキットで、オリジナルのプロトコルにしたがって逆転写を行った。
■ 2-2. FastGene™ Scriptase Ⅱ cDNA Synthesis
■ 2-3. I 社
■ 2-4. T社
すべてのキットで、オリジナルのプロトコルにしたがって逆転写を行った。
• RNAのインプット量:total RNA 500ng
• プライマー:oligo dT
• 逆転写後のRNaseH反応:なし
• プライマー:oligo dT
• 逆転写後のRNaseH反応:なし
 それぞれのキットで逆転写反応後、 PCR grade water(滅菌した水)で5 倍希釈を行い、cDNA とした
それぞれのキットで逆転写反応後、 PCR grade water(滅菌した水)で5 倍希釈を行い、cDNA とした|
PCR条件
|
|||
| ●反応組成 Takara GXL buffer 2 μL Takara dNTP mixture 0.8 μL Takara primerstar GXL 0.2 μL cDNA 1 μL Forward Primer(2 μM) 1 μL Reverse Primer(2 μM) 1 μL PCR grade water 4 μL total 10 μL |
● PCR プログラム Denature 98℃ 10sec Annealing 55℃ 15sec Extension 68℃ 1min |
|
● Primer セットについて Gene1:サイクリンB 遺伝子(約1 kb) Gene2:転写因子遺伝子(約1 kb) Gene3:受容体遺伝子(約3 kb) |
|
電気泳動
|
|||
| PCR サンプル : 3μL ゲル : 0.8% アガロースゲル 電気泳動条件:100V /20min |
RNA抽出 – 結果
① 測定機器:NanoDrop™とQubit™(Thermo Scientific)
| Sample# | Kit | Material | Starting amount (mg) |
Elution (μL) |
Yield (μL) |
Nanodrop™ | Qubit™ | RNA収量 (μg) |
|
| conc.RNA (ng/μL) |
260/280 | conc.RNA (ng/μL) |
|||||||
| 1 | Q社(植物用) | 5 shoots | 10.9 | 30 | 28.0 | 394 | 2.18 | 255 | 7.140 |
| 2 | Q社(植物用) | 5 shoots | 10.4 | 30 | 27.0 | 338.9 | 2.20 | 231 | 6.237 |
| 3 | FG | 5 shoots | 8.6 | 30 | 27.4 | 72.0 | 2.02 | 53 | 1.452 |
| 4 | FG | 5 shoots | 7.4 | 30 | 29.0 | 75.0 | 2.03 | 57 | 1.653 |
| 5 | FG mod | 5 shoots | 8.6 | 30 | 28.4 | 203.5 | 2.09 | 149 | 4.146 |
| 6 | FG mod | 5 shoots | 7.5 | 30 | 29.0 | 178.2 | 2.07 | 136 | 3.944 |
| *Q 社:Q社キット(植物RNA抽出キット) *FG : FastGene™ RNA Premium Kit (original large-input protocol) *FG・mod: FastGene™ RNA Premium Kit (modified large-input protocol) *Starting amount(mg):RNA抽出時の組織重量 *Yield(μL):NanoDrop™ に入力される値、実際の溶出量 *RNA収量(μg):Qubit™ conc× Yield |
|||||||||
●FastGene™ kit のオリジナルプロトコルでは植物の組織がFilter columnを十分に通過できないためか、収量の低下がみられた。
●Filtering をQIA shredderに変更することにより収量の改善がみられた。
●Filtering をQIA shredderに変更することにより収量の改善がみられた。
② 測定機器 :Bioanalyzer (Agilent Techonologies)
測定キット :Agilent RNA 6000 nano kit(Agilent Techonologies)
測定キット :Agilent RNA 6000 nano kit(Agilent Techonologies)
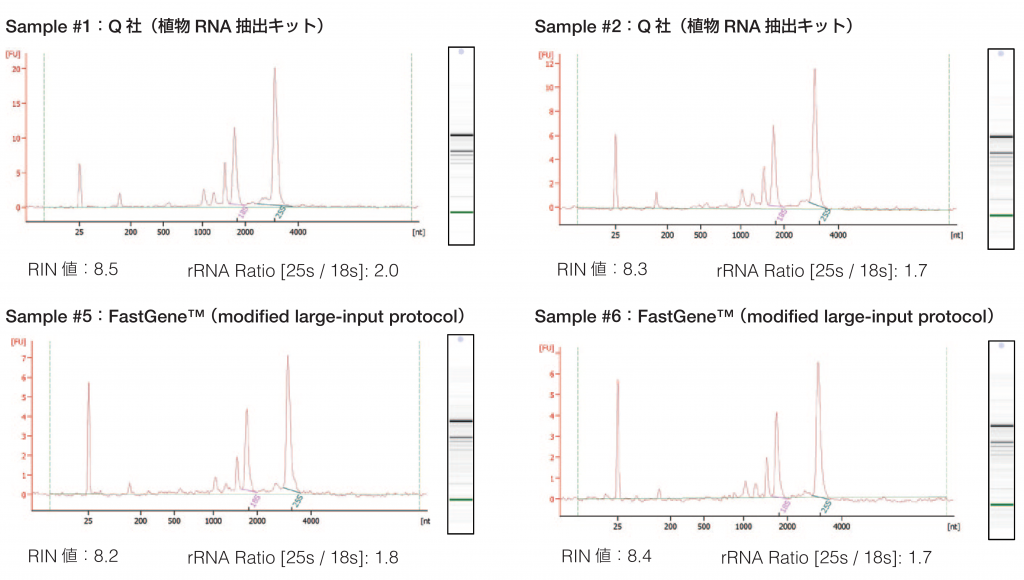
●modified protocolで得られたRNAのクオリティは問題なかった。
● RNA 抽出結果
modified large-input protocolではQ社のキットに比べ収量がやや低かった。DNase 処理のため一度溶出するプロトコルが原因と考 えられるが、遺伝子クローニングやリアルタイムPCRを目的とした逆転写反応や、NGSによるRNA-seqのライブラリを作成するのに 十分な量、クオリティのRNAを抽出することができた。
modified large-input protocolではQ社のキットに比べ収量がやや低かった。DNase 処理のため一度溶出するプロトコルが原因と考 えられるが、遺伝子クローニングやリアルタイムPCRを目的とした逆転写反応や、NGSによるRNA-seqのライブラリを作成するのに 十分な量、クオリティのRNAを抽出することができた。
逆転写反応 – 結果
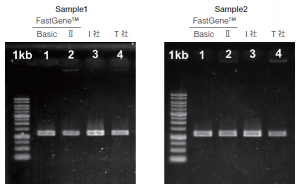
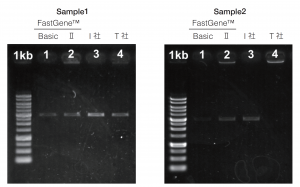
| [レーン情報] 1kb Ladder Marker 逆転写反応キット 1 FastGene™ Scriptase Basic 2 FastGene™ ScriptaseⅡ 3 I 社 キット 4 T 社 キット |
 |
|
| Gene1:サイクリンB 遺伝子(約1 kb) |
||
●サイクリンのようにシュート頂組織で高発現な遺伝子については、本実験では違いがみられなかった。
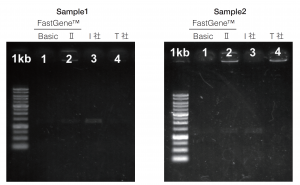
| Gene2:転写因子遺伝子(約1kb) |
 |
|
●FastGene™ ScriptaseⅡ は転写因子のように発現量の低い遺伝子でも高効率で逆転写ができているようだった。今回はPCRサイクル数を30としたが、サイクル数を増やせば十分量の産物が得られると考えられる。Basic Kitでもわずかに増幅がみられた。
| Gene3:受容体遺伝子(約3kb) |
 |
|
●3 kbのcdsを持つ受容体遺伝子も問題なく増幅できた
● 逆転写反応結果
• 全体としてI 社が良い結果になったが、FastGene™ ScriptaseⅡ cDNA Synthesisでも良い結果を得ることができた。
• PCRの結果からはゲノムからの増幅がみられなかったため、FastGene™ RNA Premium Kitによる抽出でゲノムのコンタミが十分に 除去されていることが示唆された。
• 全体としてI 社が良い結果になったが、FastGene™ ScriptaseⅡ cDNA Synthesisでも良い結果を得ることができた。
• PCRの結果からはゲノムからの増幅がみられなかったため、FastGene™ RNA Premium Kitによる抽出でゲノムのコンタミが十分に 除去されていることが示唆された。

本資料のRNA抽出について ~日本ジェネティクス(株)より
「植物」は多糖類などを多く含むため、植物の種類やその生長ステージ、更には部位(葉、茎、根、種子など)によっても大きく異なりますが、一般的にRNAの抽出難易度が高いサンプルです。このため、バッファーなどを最適化した植物専用RNA抽出キットが市販されています。
本資料で使用されているFastGene™ RNA Premium Kit は、動物細胞や組織からのRNA抽出用に開発されたキットです。
そのため、「植物」からのRNA抽出は、本キットのサポート対象外となります。
しかしながら、今回、古賀様のご厚意により、『植物サンプルにおいても他社の製品と組み合わせることで、ダウンストリームアプリケーションでの必要十分量の回収量を得ることができた事例』をご紹介させていただきましたので、ご参照ください。
- こちらのアプリケーションノートのPDFダウンロード : こちら
- 製品情報詳細ページ: FastGene™ RNA Premium Kit:
: FastGene™ Scriptase Basic cDNA Synthesis
: FastGene™ Scriptase Ⅱ cDNA Synthesis
- アプリケーションノート検索ページ(型番・キーワード・アプリケーションから検索可能)




















FastGene™ RNA Premium kit は、比較的多量の組織からごく少量の組織まで、単一のキットでカバーできる点や、少量の場合は最終溶出の容量を10μLまで減らせる点が使いやすく感じています。
また植物はイントロン配列が短いことからゲノムDNAのコンタミが問題になりやすいですが、本キットでは溶液中でDNase反応を行なうことで効果的にDNAの除去ができていると感じました。
逆転写酵素についても、長い配列をもつ遺伝子でもしっかりと逆転写できており、クローニング等の実験においては十分な品質であると感じました。
いずれも使用感や品質はこれまで使っていたキットと遜色がないですが、低価格なためとても使いやすいキットです。