日本ジェネティクスのアプリケーションノートとは?
- 当社製品を実際にご使用頂いた、正真正銘、日本国内の研究者様による評価データ
- 製品をご検討中の方はもちろん、すでにお使いのお客様におかれましても、類似の研究をされている他の研究者の方の事例集としてご活用頂けます
アプリケーション検索専用ページはこちら
メーカー名:KAPA BIOSYSTEMS 社
下記のデータは、理化学研究所情報基盤センター バイオインフォマティクス研究開発ユニット 梅田 茉奈 様、林 哲太郎 様、笹川 洋平 様、二階堂 愛 様のご厚意により掲載させていただきました。
<研究室URL:http://bit.riken.jp/>
はじめに
KAPA RNA HyperPrep Kitシリーズ*は、従来のstranded RNA-Seqライブラリー調製キットで実施される複数の酵素反応ステップを統合し、ワークフローをstreamline化した新しいキットです。
(*「mRNAキャプチャー」や「rRNA除去(対象:ヒト、ラット、マウス)」モジュールが付属したキットも提供されております。)
このように、ワークフローのstreamline化で「操作の煩雑性」や「精製によるサンプルロスのリスク」を低減させることにより、これまでよりも短時間の操作で、再現性高く、ストランド特異的な発現解析が可能となります。
今回は、効果的なstranded RNA-Seq用ライブラリー調製キットの選定を目的として、KAPA RNA HyperPrep Kit with RiboEraseを用い、マウスEmbryonic stem cell(ES cell)と、そこから分化させたPrimitive endoderm cell(PrE cell)についてそれぞれrRNA depletion(rRNA除去)RNA-Seqを行い、評価した事例についてご紹介します。
評価デザイン
●RNA抽出 ⇒ QC(結果①)
↓
●rRNA depleted RNA ⇒ QC(結果②)
↓
●ライブラリー調製 ⇒ QC(結果③)
↓
●シーケンス
↓
●データ解析 ⇒ rRNA除去効率(結果④)
カバレッジ(結果⑤)
再現性(結果⑥)
マーカー遺伝子の解析(結果⑦)
• 従来よりも酵素反応と精製のステップを最小化することでライブラリー 調製操作を省力化
• 短時間(約4時間)でライブラリー調製が可能
• 以下のモジュール付属キットも提供可
・mRNAキャプチャー
・rRNA除去(ヒト、ラット、マウス)
注:アダプターは含まれておりません。詳細はお問い合わせください。
実験条件
• 初発サンプル (細胞) : マウスEmbryonic stem cell(ES cell)
マウスPrimitive endoderm cell(PrE cell)
*PrE cellは、ES cellをデキサメタゾンで72hr. 処理して誘導、分化させた。
• RNA抽出方法 : 1.0×106 個の細胞をQIAzol Lysis Reagent(Qiagen)で溶解し、Direct-zolTM RNA MiniPrep Kit( Zymo Research)でDNase I処理も行ってからtotal RNAを抽出した。
• 抽出したRNAの確認 : 品質 Bioanalyzer RIN >9.5、濃度測定 Nano-drop
• ライブラリー調製キット : KAPA RNA HyperPrep Kit with RiboErase
*全てプロトコール推奨反応量の1/2量で実施
• インプットRNAの調製 : i. ES cell total RNA
ii. ES cell poly(A)selected RNA(*)
iii. PrE cell total RNA
iv. PrE cell poly(A)selected RNA(*)
*今後の検証のため、NEB社キットでpoly(A)RNA を精製したサンプルも調製した。条件を揃えるためrRNA除去ステップから行った。
• インプットRNA量 : i ~ iv 各200ng, n=3
*poly(A)selected RNAサンプルについては、total RNA 200 ng 換算量を用いた。
• 外部評価用 spike RNA : ERCC RNA Spike-In Mix(ThermoFisher)
*推奨条件で添加(1ngあたり0.00001倍希釈したものを2μL添加)
*poly(A) selected RNAサンプルにおいても、total RNA 200 ng 換算で添加
• RNA断片化条件 : 85℃ 6min(目標サイズ:300-400 bp)
• 使用アダプター : SeqCap Adapter Kit (Roche)
• アダプター添加濃度 : 1.5 μM
• ライブラリー増幅サイクル数 :7~8cycle
• シーケンサー :HiSeq 2500
KAPA RNA HyperPrep Kit with RiboEraseのワークフロー
KAPA RNA HyperPrep Kit with RiboErase (HMR)
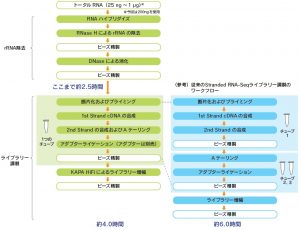
従来のstranded RNA-Seqライブラリー調製キットで実施される複数の酵素反応ステップが統合され、ワークフローがstreamline化されている。
結果
① 抽出したRNAの品質(一部)(Agilent Bioanalyzer RNA 6000 Nano Kit)
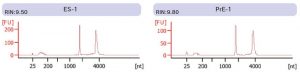
全てのサンプルでRIN>9.50であることを確認した。
②-1 rRNA 除去後のRNAの品質(Agilent Bioanalyzer RNA 6000 Pico Kit)
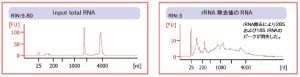
②-2 rRNA 除去後のRNAの品質(qPCRによる特定遺伝子の定量)
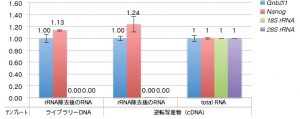
※ Gnb2l1 の検出量で補正した値を用いて total RNA の逆転写産物を1とした時の相対値
rRNA 除去後のRNA では、qPCR で28S および18S rRNA は検出されなかった。
マウスES cell から抽出したtotal RNA 200ng を インプットRNAとして使用し、他社キットでrRNA 除去から2nd strand synthesis まで行った後、KAPA HyperPrep Kit( DNA-Seq用)を用いてライブラリーを作製した。
何回か実施したが、下記のデータのとおり、rRNA 除去後のQC において、想定どおりの品質が得られなかった。
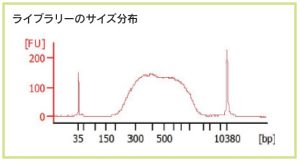
そこからライブラリー作製も試みたが、結果的に意図したサイズ分布を示さなかった。
その後、同様のサンプルを用いてKAPA RNA HyperPrep Kit with RiboErase を評価したところ、安定してライブラリーを得ることができた。
rRNA 除去後のRNA の品質(qPCR による特定遺伝子の定量)
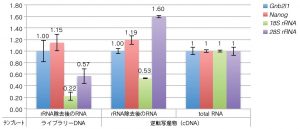
※ Gnb2l1 の検出量で補正した値を用いて total RNA の逆転写産物を1とした時の相対値
rRNA 除去後のRNA も、qPCR で28Sおよび18S rRNA が検出された。この結果から、rRNA の除去が不十分であることが示唆された。
作製したシーケンスライブラリDNA の品質
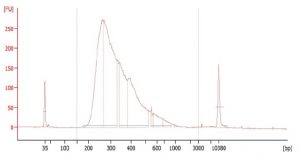
ライブラリDNA のサイズ分布を確認したところ、意図とは異なるサイズ分布を示した。
| Library DNA の収量 | 平均 (ng) |
標準偏差 (ng) |
PCR cycle |
|---|---|---|---|
| i. ES cell total RNA | 59.92 | 5.70 | 7 |
| ii. ES cell poly(A)selected RNA | 48.13 | 3.79 | 8 |
| iii. PrE cell total RNA | 61.33 | 4.44 | 7 |
| iv. PrE cell poly(A)selected RNA | 47.27 | 4.89 | 8 |
| (参照)別途実施ES cell total RNA | 41.64 | 1.73 | 6 |
※各n=3
得られたシーケンスリードについて、rRNAのReferenceデータを参照としてrRNA由来リードの混入率を検証したところ、rRNAが効果的に除去され、十分に低いことが確認された。
<RepeatMasker repClass: rRNA>
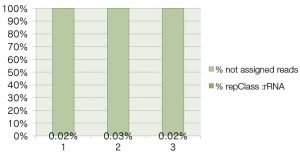
RepeatMasker のrepClass: rRNA (total 1564 sites)をReferenceに用いて計算したもの。この計算方法では、rRNAはほとんど除去されている。
<RefSeqGene: Rn45s or Rn4.5s>
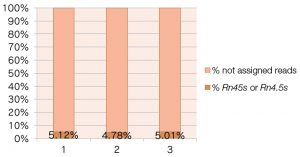
RefSeqGeneのRn45s(45S pre-ribosomal RNA)と Rn4.5s(4.5S RNA)をReferenceに用いて計算したもの。この計算方法では、約5%のrRNAの混入が認められる。
<rRNA 除去を実施したRNA-Seq のリードの構成>
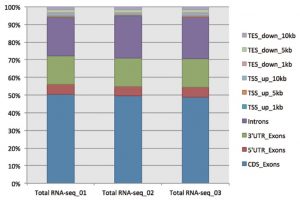
シーケンスリードの構成を調べたところ、リードの20%以上をIntrons(■紫)が占めており、poly(A)型だけでなく、非poly(A)型RNAも効率良くシーケンスされていることが確認された。
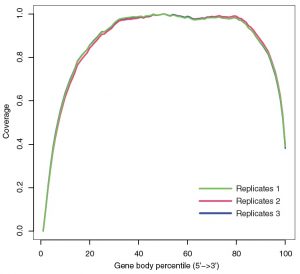
⑤ カバレッジ
遺伝子構造を確認するのに重要な「トランスクリプトのカバレッジ」が非常に均一であった。
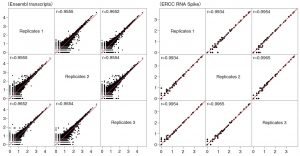
⑥ 再現性
発現量の散布図から、遺伝子発現の十分な再現性が認められた。ERCC spikeなど外部評価RNAでも十分な相関が有ることが確認された。
⑦ マーカ遺伝子の解析
実際にゲノムビューワーで確認した。RNA-Seqの結果として、ES細胞、PrE細胞それぞれの細胞に特異的なマーカー遺伝子が確認された。



- こちらのアプリケーションノートのPDFダウンロード : こちら
- アプリケーションノート検索ページ(型番・キーワード・アプリケーションから検索可能)
<2018年12月までの期間限定キャンペーン実施中!>



















RNaseH 方式による rRNA-depletion では、RNA 溶液を 45℃ に保ったまま次の試薬を加えて混ぜる必要があるのですが、この過程は比較的煩雑に感じました。
しかし、再現性の有る rRNA-depletion が行えたのでとても満足しました。
Poly-A RNA-seq だと上述のような煩雑な過程を必要としないため、さらに簡便です。実際に操作を行うにあたり、注意点が詳細にプロトコルに書かれていますので、問題は特に感じずプロトコル通りに進めるだけで問題ないと感じました。十分な精度が安定して出ているため、 rRNA-depletion 方式の RNA-seq kit としてファーストチョイスになると思います。